Аннотация
Западные медоносные пчелы (Apis mellifera) являются одними из наиболее важных опылителей сельскохозяйственных культур и дикорастущих растений. Несмотря на рост доступности данных о последовательности медоносных пчел, филогения вида остается предметом споров. В частности, географическое происхождение медоносных пчел остается неопределенным, как и взаимоотношения между составляющими их родами и подвидами. Мы стремимся выяснить эволюционную и биогеографическую историю медоносной пчелы на основе митохондриальных геномов. Здесь мы анализируем полные митохондриальные геномы 18 подвидов A. mellifera, принадлежащих ко всем основным линиям, используя ряд стратегий выборки генов и моделей вывода, чтобы определить факторы, которые могли способствовать получению несоответствующих результатов в предыдущих исследованиях. Наш анализ подтверждает североафриканское или ближневосточное происхождение A. mellifera. Мы показываем, что ранее предполагаемые европейская и афротропическая колыбели медоносных пчел являются результатом филогенетической ошибки. Монофилия линий M, C и O сильно поддерживается, но линия A представляется парафилетической. A. mellifera колонизировала Европу по крайней мере двумя путями — через Гибралтарский пролив и через Малую Азию.
Введение
Поскольку Apis mellifera Linnaeus, 1758 является, вероятно, самым важным опылителем сельскохозяйственных культур и диких растений, а также производителем разнообразных пищевых продуктов, имеющих пищевое, медицинское и косметическое назначение, таких как мед и прополис, важность понимания эволюционной истории Apis mellifera невозможно переоценить. Хотя западная медоносная пчела родом из Европы, Африки, Ближнего Востока и некоторых районов Азии (1), способность A. mellifera колонизировать практически все пригодные для жизни биомы на Земле и адаптироваться к различным биоклиматическим условиям является живым доказательством удивительной морфологической и поведенческой пластичности этого вида (2). На сегодняшний день описано более 30 отдельных подвидов (или «географических рас») (3,4,5,6,7). Различия между подвидами во многих случаях не очевидны при осмотре живых или приколотых особей, и идентификацию приходится проводить на основе количественных морфометрических (6,8,9) или молекулярных анализов (5,10,11). Классификация подвидов медоносной пчелы имеет важное практическое значение для пчеловодства. Пчеловоды давно признали, что расы пчел отличаются по ряду поведенческих признаков, таких как спокойствие, интенсивность роения, производство меда, способность использовать различные источники корма и устойчивость к болезням (6,12,13,14,15). Панконтинентальное исследование европейских подвидов показало, что адаптированные к местным условиям особи имеют более высокую выживаемость и более низкий уровень патогенов (16,17), что подчеркивает важность сохранения генетического разнообразия местных популяций медоносных пчел (7,18).
Надежная магистральная филогения западной медоносной пчелы позволит апидологам изучить эволюцию экономически важных признаков у этого вида и основы его адаптации к различным условиям окружающей среды (19). Хотя в настоящее время доступно больше генных последовательностей из всего ареала A. mellifera, чем когда-либо ранее (20), на ряд давних вопросов до сих пор нет ответа. В частности, географическое происхождение западной медоносной пчелы исторически было предметом споров (1,21,22). Последние результаты анализа целых геномов подтверждают либо северо-восточное африканское, либо ближневосточное происхождение A. mellifera (23), но различить эти две гипотезы с помощью любого набора данных оказалось непросто. Напротив, анализ полных митохондриальных геномов показал, что A. m. mellifera является самым базальным подвидом, что позволяет отнести происхождение медоносных пчел к Северной Европе (24,25,26,27,28,29,30). Европейское происхождение медоносных пчел не кажется необоснованным, поскольку самые древние однозначные ископаемые представители рода Apis известны из олигоцена Франции и Германии, а ископаемые европейские медоносные пчелы также демонстрируют высокую степень морфологического разброса (4,31,32,33,34). С другой стороны, учитывая, что все остальные ныне живущие виды рода Apis встречаются в Азии, идея о том, что A. mellifera могла первоначально расселиться с востока, пользовалась устойчивой популярностью с 1950-х годов (1,35,36,37,38). Неожиданно, поддержка происхождения медоносных пчел из тропической Африки была получена в результате анализа 1 136 SNPs у 341 пчелы (2). Примечательно, что в большинстве последних филогеномных исследований, посвященных происхождению медоносных пчел (1,2,39), использовался алгоритм объединения соседей (NJ), который подвержен систематическим ошибкам и может давать вводящие в заблуждение топологии (40).
Взаимоотношения между отдельными подвидами A. mellifera также неясны, а исследования дают неоднозначные результаты. Традиционно западная медоносная пчела делится на четыре эволюционные линии: линия А (подвид, родом из Африки), линия М (Западная и Северная Европа), линия С (Южная и Восточная Европа) и линия О (Кавказ, Турция, Ближний Восток, Кипр, Крит) на основе морфометрических и молекулярных данных (2,6,41,42,43). В последние годы на основе молекулярных данных были предложены две новые, предположительно изолированные линии. Медоносные пчелы из Эфиопии значительно отличаются от линии А, в которую они были первоначально помещены, и поэтому были отнесены к отдельной группе, линии Y (44). Шестой род из Сирии и Ливана был идентифицирован как явно дивергентный от рода О на основе соседства микросателлитных локусов и митохондриальной ДНК (45,46). Здесь мы называем эту предполагаемую группу линией S. Более того, некоторые исследования не признают различий между линиями С и О (21,38,47).
Чтобы пролить свет на противоречивые взаимоотношения между подвидами медоносной пчелы и обеспечить надежную базовую филогению A. mellifera, мы использовали коллекцию недавно секвенированных полных митохондриальных геномов, представляющих более половины описанных подвидов медоносной пчелы со всего ареала ее обитания. Мы проанализировали наши данные с помощью методов, позволяющих выявить источники филогенетической несогласованности, используя различные стратегии выборки генов и модели вывода.
Материал и методы
Данные секвенирования
Митохондриальный геном A. mellifera состоит примерно из 16 463 п.н. и включает 13 генов, кодирующих белки, 22 гена трансферной РНК (тРНК), два рибосомальных гена (рРНК) и один контрольный регион (48). Все митохондриальные геномы A. mellifera, секвенированные на сегодняшний день, были получены из GenBank в январе 2020 года вместе с митохондриальными геномами A. cerana, A. florea и A. dorsata, которые были использованы в качестве аутгрупп. Всего было проанализировано 18 митогеномов A. mellifera, представляющих более половины ее известного подвидового разнообразия. Номера доступа GenBank приведены в таблице S1
Данные для 13 белок-кодирующих генов и двух молекул РНК были загружены, выровнены и конкатенированы с помощью PHYLOSUITE v1.2.1 (49). Белково-кодирующие гены были однозначно выровнены благодаря небольшому количеству пробелов и их структуре на основе кодонов с помощью алгоритма G-INS-i, реализованного в плагине MAFFT v 7.313 (50). Рибосомные РНК 16S и 18S были выровнены в MAFFT с помощью алгоритма E-INS-I.
Филогеномные анализы
Чтобы проверить влияние выборки генов на восстановление топологии, мы подготовили три набора данных: только первая и вторая позиции кодонов (P12), P12 и два гена рРНК (P12RNA) и все позиции кодонов вместе (P123). Было показано, что третья кодоновая позиция генов, кодирующих белки, характеризуется высокой степенью насыщенности, что потенциально может привести к смещенным филогениям, и поэтому ее исключение рекомендуется для снижения гетерогенности данных (51,52).
Для изучения потенциальных источников филогенетического конфликта, возникающего в результате неправильного выбора модели, мы проанализировали последовательности, используя как сайт-гетерогенный Байесовский вывод (BI), так и сайт-гомогенный метод максимального правдоподобия (ML). Неправильный выбор моделей вывода представляет собой один из ключевых источников филогеномных ошибок (53). В частности, высокая композиционная и скоростная гетерогенность последовательностей может привести к тому, что более простые сайт-однородные модели дают результаты, подверженные систематическим ошибкам, таким как притяжение длинных ветвей (54,55). Такие типы ошибок часто трудно заметить, потому что они могут быть сильно поддержаны и последовательно восстановлены (56). С другой стороны, более сложные сайт-гетерогенные модели, учитывающие композиционную и скоростную неоднородность, обычно лучше соответствуют данным и поэтому применяются для решения сложных филогеномных проблем, таких как происхождение эукариот или базальный порядок ветвления в Metazoa (54,55,57,58,59,60,61,62,63).
Модель гетерогенной смеси сайтов BI CAT-GTR + G была запущена в PHYLOBAYES MPI 1.7 (64). Две независимые цепи Марковской цепи Монте-Карло (MCMC) запускались до сходимости (maxdiff < 0.3). Для каждого прогона PHYLOBAYES мы использовали программу bpcomp для получения результатов наибольшего (maxdiff) и среднего (meandiff) расхождения, наблюдаемого во всех бипартициях.
Для ML-анализа объединенные наборы данных, разбитые по кодонам, были проанализированы с помощью программы PARTITIONFINDER 2.1.1 для выбора наиболее подходящих моделей (65). Для поиска «всех» моделей по Байесовскому информационному критерию использовался «жадный» алгоритм, при этом длины ветвей не связывались. Схема разбиения представлена в таблице S2. ML-анализ с использованием выбранных моделей был выполнен в IQ-TREE v 1.6.12. Анализы проводились с использованием 1 000 сверхбыстрых бутстрапов66.
Все иллюстрации были подготовлены с помощью iTOL v. 5.5.1 (67), Adobe Photoshop v. 21.2 и Adobe Illustrator v. 24.2.
Результаты
Наибольшее расхождение (maxdiff) во всех прогонах PHYLOBAYES было < 0,3, что указывает на то, что все они представляют собой филогенетически информативные прогоны (68). Деревья, восстановленные с помощью модели CAT-GTR + G, представлены на рис. 1, деревья с вычисленными длинами ветвей и топологии, восстановленные с помощью сайто-однородных ML моделей, представлены на рис. S1-S12. Филогенетическая гипотеза, основанная на CAT-GTR + G анализе P12RNA, представлена в виде графической аннотации. Неглубокие связи между подвидами обычно получали высокую поддержку, хотя то же самое не всегда было характерно для более глубоких узлов, как это было типично для большинства филогений медоносной пчелы, проведенных на сегодняшний день (7,20).
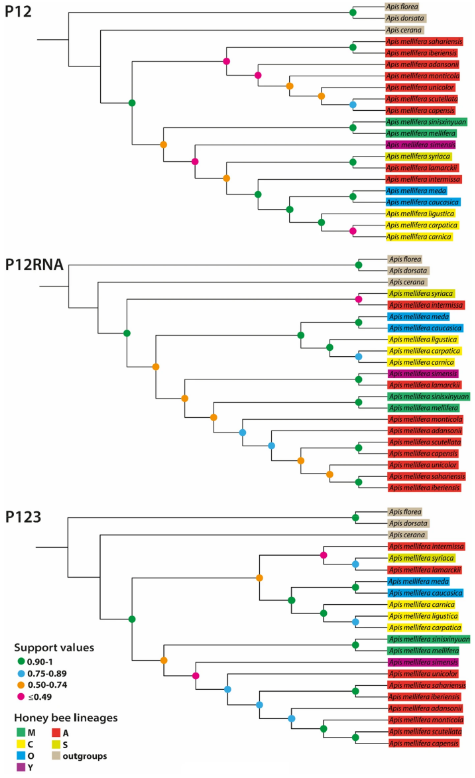
Наши анализы с использованием сайт-гетерогенной модели CAT-GTR + G всегда восстанавливали подвиды из северной Африки и Ближнего Востока как сестринские по отношению к остальным подвидам A. mellifera. Полная поддержка (Bayesian Posterior Probabilities [BPP] = 1) была получена для набора данных P12RNA, где A. m. syriaca и A. m. intermissa образовали самый нижний кладезь. Первый вид является родным для восточного Средиземноморья, а второй встречается в Тунисе, Алжире и Марокко между Атласскими горами и побережьями Средиземного моря и Атлантического океана (69,70). Анализ наборов данных P12 и P123 показал, что A. m. sahariensis (северо-западная Африка) + A. m. iberiensis (Пиренейский полуостров) и A. m. syriaca (Ближний Восток и Израиль) + A. m. intermissa (северная Африка) являются самыми базальными подвидами, соответственно, хотя и с низкой поддержкой.
ML-анализ P12 и P12РНК подтвердил, что североафриканский A. m. intermissa является самым базальным подвидом с полной поддержкой (Maximum Likelihood Bootstrap [MLB] = 100, рис. S2, S4). Клад, включающий A. m. mellifera и A. m. sinisxinyuan, оказался сестринским по отношению к остальным медоносным пчелам в ML-анализе P123 (MLB = 100, рис. S6).
Независимо от анализируемого набора данных или используемой модели, монофилия рода M всегда подтверждалась с максимальной поддержкой (BPP = 1, MLB = 100). Аналогичным образом, линии C и O всегда были монофилетичными и вместе образовывали монофилетичную кладу. Примечательно, что линия A никогда не была монофилетической в анализах PHYLOBAYES, что привело к разделению между базальными северными африканскими подвидами и более производными афротропическими.
Обсуждение
Колыбель западных медоносных пчел: Азия, Африка, Ближний Восток или Европа?
Разъяснение филогении A. mellifera долгое время было затруднено ограниченной доступностью молекулярных данных для большинства подвидов медоносной пчелы. Молекулярные анализы традиционно выявляют противоречивые отношения между расами медоносных пчел, что приводит к неопределенности в отношении географического происхождения вида (1).
Азиатское происхождение медоносных пчел кажется довольно интуитивным, и, вероятно, именно по этой причине оно уже давно пользуется поддержкой в апидологических кругах (1,35,36,37). Из десяти или около того признанных видов рода Apis все, за исключением A. mellifera, встречаются в Азии (71), поэтому было бы разумно ожидать, что этот регион также является историческим центром западной медоносной пчелы. Родной ареал A. mellifera в Азии включает Казахстан, Монголию, юг России, а отдельный подвид A. mellifera также встречается в Китае (3,72). Несмотря на то, что в ходе последних геномных анализов медоносных пчел не удалось обнаружить азиатскую колыбель западной медоносной пчелы, это объяснение по-прежнему считается наиболее правдоподобным (1,39); в конце концов, базальные азиатские популяции могли просто вымереть или еще не были секвенированы. Однако мы предупреждаем, что эволюционная история рода Apis может быть значительно сложнее. Хотя самые древние представители рода Apis известны из олигоцена Западной Европы, эти виды не обязательно являются базальными и морфологически похожи на сохранившуюся гигантскую медоносную пчелу A. dorsata, обитающую в Южной и Юго-Восточной Азии (32,33,34). Это указывает на то, что род Apis мог существовать в Европе задолго до появления A. mellifera, и поэтому нет причин ожидать азиатского происхождения этого вида.
Гипотеза о происхождении медоносной пчелы из тропической или субтропической Африки появилась относительно недавно и была предложена в первом SNP-анализе взаимоотношений медоносных пчел, проведенном Уитфилдом и др. (2). Однако обширные повторные анализы исходного набора данных Уитфилда и коллег, исключающие образцы с потенциально гибридным происхождением (1) , а также анализ новых SNPs (39) не нашли однозначной поддержки гипотезы «из Африки». Примечательно, что во всех трех исследованиях (1,2,39) использовался алгоритм соседнего соединения (NJ) для вывода эволюционных отношений. Хотя NJ отличается высокой скоростью вычислений и может быть использован для получения быстрого справочного дерева, он также подвержен систематическим ошибкам (40) и поэтому не рекомендуется для филогенетических исследований (56). Анализы, в которых использовались методы вывода ML, отвергли глубокое африканское происхождение A. mellifera (23). Наш анализ с использованием модели CAT-GTR + G, которая была разработана специально для борьбы с эффектами систематической ошибки, также не подтвердил афротропическое происхождение западных медоносных пчел. Поэтому кажется, что результат «из Африки» является филогенетическим артефактом, вероятно, вызванным систематической ошибкой.
Наш анализ показал, что северные африканские подвиды, а иногда и ближневосточные A. m. syriaca, являются самыми низкорослыми западными медоносными пчелами. Этот результат согласуется с крупнейшей молекулярной филогенией, основанной на целых геномах медоносных пчел со всего ареала вида (23) , и с классическим морфометрическим анализом 33 признаков 404 пчел, проведенным Раттнером и др. (21) . Однако этот вывод противоречит результатам ML-анализа митохондриальных геномов медоносной пчелы, в результате которого темная европейская медоносная пчела (A. m. mellifera, линия M) была признана самым нижним подвидом. В анализах, которые выявили европейскую колыбель A. mellifera, неизменно использовались однородные модели (24,25,26,27,28,29,30). В нашем анализе мы восстановили линию M как самую базальную группу только при анализе набора данных P123 в рамках ML. Тот же результат не был получен при ML-анализе наборов данных P12 и P12RNA без учета неоднородного положения третьего кодона, а также при анализе данных с помощью модели CAT-GTR + G. Поскольку европейское происхождение медоносных пчел восстанавливается только в анализах, которые не учитывают смещающее влияние композиционной неоднородности данных, мы пришли к выводу, что оно, скорее всего, является артефактным.
В целом, происхождение A. mellifera, по-видимому, лежит в северной Африке или на Ближнем Востоке. Различать эти два региона на современном этапе сложно, поскольку секвенировано лишь несколько митохондриальных геномов местных рас пчел. Сестринские отношения между африканской A. m. intermissa и ближневосточной A. m. syriaca, которые были восстановлены как самый базальный кладе в нашем CAT-GTR + G анализе P12RNA, позволяют предположить, что предковый ареал A. mellifera, возможно, охватывал оба региона. Поэтому, чтобы сузить географическое происхождение западной медоносной пчелы, необходимы данные по большему количеству подвидов, встречающихся в этом регионе.
Взаимоотношения между подвидами Apis mellifera
Учитывая скудное количество морфологических различий между подвидами медоносной пчелы, исторически сложилось так, что их определяли в основном по количественным измерениям около 40 основных признаков, таких как окраска, волосистость, характер крыльев и размеры различных частей тела6. К сожалению, количественные морфологические признаки трудно поддаются филогенетическому анализу (73) и страдают от широко распространенной гомоплазии (74). Тем не менее, наш анализ выявил сильную поддержку трех из четырех морфологически определенных родов медоносных пчел: Линии O, C и M. Линии O и C образуют монофилетическую группу с сильной поддержкой, что уже предполагалось морфометрией (6) и более ранними анализами митохондриальных маркеров (38,74). Два подвида линии M, европейский A. m. mellifera и китайский A. m. sinisxinyuan, также были обнаружены во всех анализах. Очевидную парафилию рода А трудно интерпретировать, поскольку она не была обнаружена с высокой степенью поддержки. Тем не менее, разделение северных и южноафриканских медоносных пчел на два различных клада было ранее обнаружено при анализе ограниченного числа митохондриальных маркеров (44,75), поэтому проблема требует дальнейшего изучения.
Помимо четырех традиционных морфологических групп, молекулярные исследования предполагают существование по крайней мере двух дополнительных линий. Линия Y была признана в качестве отличительной группы медоносных пчел из Эфиопии на основе анализа парсимонии микросателлитных маркеров и митохондриальной ДНК (44). Отличительные особенности эфиопских пчел, традиционно относимых к линии А, были также продемонстрированы феромонными (76) и морфометрическими анализами (77,78), хотя последние также обнаружили высокую степень интрогрессии между эфиопскими популяциями и соседними четко определенными африканскими подвидами. Мы обнаружили тесную близость между эфиопским A. m. simensis и частью парафилетической линии A, четко отделяя его от географически близкой линии O. Эти результаты согласуются с ML-анализом Кридланда и др. (23). Филогенетическое положение A. m. simensis было слабо поддержано или не поддержано вообще в наших анализах CAT-GTR + G. Поэтому в настоящее время нельзя отвергать обоснованность выделения A. m. simensis в отдельный род, но это зависит от будущего подтверждения.
Род S, состоящий из A. m. syriaca, был рассмотрен как отдельный род пчел Альбураки и др. (46). Этот подвид встречается на территории Сирии, Ливана и северной Иордании и был отнесен к роду A на основании морфологических признаков (6,21), хотя митохондриальный анализ поместил его в род O (42). Положение сирийской медоносной пчелы было восстановлено без поддержки или с незначительной поддержкой, хотя родственные отношения с A. m. lamarckii были обнаружены при анализе CAT-GTR + G набора данных P12 (BPP = 0,93, рис. 1). Таким образом, вопрос о филогенетическом положении сирийской пчелы остается открытым.
Биогеографическая история западных медоносных пчел
Независимо от анализируемого набора данных, наш байесовский анализ последовательно подтвердил наличие родственных связей между близко встречающимися A. m. sahariensis, обитающими в оазисных регионах пустыни Сахара, и A. m. iberiensis из Испании и Португалии (рис. 1). Иберийская медоносная пчела была вложена в линию А в наших анализах независимо от анализируемого набора данных. Хотя она была помещена в род М на основании морфологических признаков (6,79,80), молекулярные анализы показывают, что эта группа имеет значительную степень смешения с родом А, особенно на юге Пиренейского полуострова, и поэтому она была отнесена к роду А некоторыми авторами (5,38,81,82). Это указывает на то, что часть предковой североафриканской популяции медоносных пчел, вероятно, колонизировала Европу через Гибралтарский пролив. Эта гипотеза была предложена морфометрическим анализом (21 , но была поставлена под сомнение ранними анализами, основанными на митохондриальных маркерах (38,74). Следует отметить, что в этих анализах использовались методы парсимонии и расстояния, которые превосходят основанные на моделях методы BI и ML (83,84). Поэтому мы считаем кросс-гибралтарское расселение из северной Африки в Европу весьма вероятным (рис. 2).
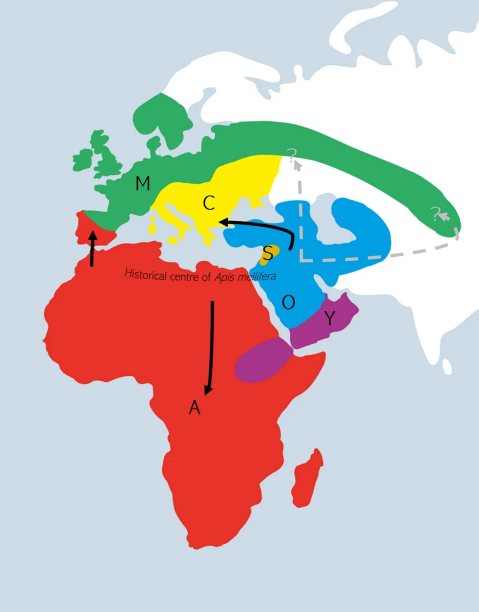
Второй маршрут миграции из северной Африки и Ближнего Востока в Европу, вероятно, проходил через Малую Азию или Кавказ. На это указывает сильно поддерживаемая клада, включающая линии O и C (рис. 1). В то время как пчелы базовой линии О встречаются на Ближнем Востоке и Кавказе, более производные пчелы линии С обитают в Южной Европе. Распространились ли медоносные пчелы в Европу через Турцию или также пересекли Кавказ и Северную Азию, еще предстоит выяснить, поскольку целые митохондриальные последовательности для филогенетически важных подвидов A. m. anatolica, A. m. macedonica и A. m. cecropia пока недоступны. Мы предварительно считаем первый вариант более вероятным, поскольку межтурецкое расселение поддерживается морфометрическими данными (21). Более того, анализ расстояния между последовательностями ND2 показал, что турецкий A. m. anatolica является сестрой ближневосточного A. m. meda (85).
Распространение медоносных пчел линии M в Европу и Азию трудно объяснить, поскольку положение линии M было восстановлено как близкое к пчелам линии A в анализах CAT-GTR + G, но без сильной поддержки. Чен и др. (3) предположили, что A. m. mellifera и A. m. sinisxinyuan близки к пчелам линий O и C на основе анализа NJ. Этот результат подразумевает, что происхождение линии М лежит в Малой Азии или на Ближнем Востоке. Очевидно, что происхождение и положение линии М требует дальнейшего изучения.
Понять, какие исторические факторы управляли расселением западных медоносных пчел, довольно сложно. Единственное однозначное свидетельство о времени эволюционных событий может быть получено из ископаемых останков. В идеале, для калибровки филогении A. mellifera (86) можно было бы использовать ископаемые останки медоносных пчел, идентифицированные до уровня подвида, что позволило бы нам соотнести события расщепления на дереве с известными биоклиматическими событиями. К сожалению, сведения о субфоссилиях A. mellifera скудны. Включения в копале часто упоминаются как самые древние субфоссилии западных медоносных пчел, но сам копал имеет неопределенное происхождение (87,88,89). Кроме того, копал, как известно, трудно датировать, и его возраст может составлять от 5 миллионов до нескольких лет (90). Другие останки медоносных пчел известны только по археологическим раскопкам, их возраст не превышает нескольких тысяч лет (91,92). Таким образом, исследования до сих пор давали весьма противоречивые результаты; разделение между A. cerana и A. mellifera произошло, по разным оценкам, от 6 до 25 миллионов лет назад (38,72,93,94), а быстрое расхождение между подвидами A. mellifera — от 0,3 до 1,3 миллиона лет назад (6,38,75). Уолберг и др. (39) использовали метод генеалогической конкордации для оценки того, что подвиды медоносной пчелы разошлись между 13 000 и 38 000 лет назад, что соответствует последнему ледниковому максимуму, подразумевая, что экспансия A. mellifera из региона ее происхождения в Европу началась после отступления ледниковых покровов. Экспансия медоносной пчелы в афротропики могла контролироваться вызванным климатом опустыниванием и сменой растительности в плейстоцене (75). Однако очевидно, что для проверки этих гипотез необходимо пересмотреть временные рамки диверсификации медоносной пчелы, имея под рукой больше ископаемых.
Будущие направления
Хотя митохондриальные геномы дают полезное представление об эволюционной истории западной медоносной пчелы, необходимо отметить несколько предостережений. Только чуть более половины признанных в настоящее время подвидов медоносной пчелы имеют свои митохондриальные геномы, а некоторые геномы из филогеографически важных регионов Ближнего Востока и Малой Азии все еще отсутствуют. Это означает, что наше понимание происхождения и распространения медоносной пчелы все еще неполно.
Низкие значения поддержки, полученные в некоторых узлах, возможно, являются результатом того, что геномы близкородственных подвидов медоносной пчелы обычно очень похожи (1,23). Бутстреп-поддержка для групп рассчитывается как доля случаев, когда данный кладе восстанавливается при повторной выборке подмножества данных (95). Таким образом, бутстреп-поддержка не обязательно отражает филогенетический сигнал, но оценивает избыточность данных, которая, как ожидается, будет низкой в последовательностях с низкой изменчивостью (96). Аналогично, байесовские апостериорные вероятности оказываются низкими при анализе последовательностей потенциально гибридного происхождения (97). Мы ожидаем, что топологии с лучшей поддержкой могут быть получены путем увеличения выборки генов в будущих анализах.
Естественная и вызванная человеком гибридизация популяций медоносных пчел привела к значительной степени генетической примеси между подвидами (98,99,100,101,102). Это относится к одичавшим и управляемым популяциям пчел в Старом Свете, а также к популяциям, завезенным в Америку и Австралию (103,104). В результате большинство подвидов медоносной пчелы не пережили длительных периодов изоляции (1) , и составленные нами эволюционные деревья представляют собой лишь прагматические приближения к эволюционной истории медоносной пчелы. В конечном счете, возможно, более уместно рассматривать эволюцию медоносной пчелы как паутину переплетающихся популяций, а не как строго дихотомическое ветвящееся дерево. Однако распутывание «эволюционной паутины» медоносной пчелы также зависит от получения большего количества целых геномных последовательностей из всего ее родного ареала.
Выводы
1.
Западная медоносная пчела произошла из Северной Африки или Ближнего Востока. Этот вывод согласуется с морфологическими (21) и наиболее обширными молекулярными (23) данными.
2.
Более ранние гипотезы об афротропном (2) или европейском (27) происхождении A. mellifera представляются ошибочными. Они никогда не восстанавливаются, когда к наборам данных применяются методы борьбы с эффектами неоднородности темпа.
3.
A. mellifera колонизировала Европу из Северной Африки или Ближнего Востока по крайней мере двумя путями: через Гибралтарский пролив и через Турцию, хотя возможный путь через Кавказ или Северную Азию подлежит проверке после секвенирования геномов подвидов, обитающих в этих регионах.
4.
Линия A была восстановлена как парафилетическая, разделенная между северо- и южноафриканскими подвидами, хотя и с низкой поддержкой.
5.
Линии O и C образуют монофилетическую группу с сильной поддержкой во всех анализах. Базовые пчелы линии O обитают на Ближнем Востоке и Кавказе, в то время как более недавно дивергировавшие пчелы линии C встречаются в Южной Европе.
6.
A. m. mellifera сгруппирована вместе с китайской A. m. sinisxinyuan. Предполагалось, что род М отделился от родов О и С в Малой Азии (3) , но наш анализ показал слабое родство с африканскими подвидами. Таким образом, происхождение линии M открыто для дальнейшего изучения.
7.
В будущих исследованиях происхождения медоносных пчел приоритетным должно быть получение образцов из северной Африки и Ближнего Востока, поскольку в этих регионах обитает большое разнообразие генетически различных подвидов (46). Эти образцы будут особенно важны для проверки монофилии линии A и положения недавно предложенных линий S и Y.
Доступность данных
Номера доступа NCBI анализируемых последовательностей указаны в Дополнительной информации. Проанализированные наборы данных и все выходные файлы доступны онлайн на сайте Mendeley Data https://data.mendeley.com/datasets/fk9whcw3pp/1 (https://doi.org/10.17632/fk9whcw3pp.1).
Дополнительная информация
Ссылки:
1.Han, F., Wallberg, A. & Webster, M. T. From where did the Western honeybee (Apis mellifera) originate?. Ecol. Evol. 2, 1949–1957 (2012). PubMed PubMed Central Google Scholar
2.Whitfield, C. W. et al. Thrice out of Africa: Ancient and recent expansions of the honey bee, Apis mellifera. Science 314, 642–645 (2006). ADS PubMed CAS Google Scholar
3.Chen, C. et al. Genomic analyses reveal demographic history and temperate adaptation of the newly discovered honey bee subspecies Apis mellifera sinisxinyuan n. ssp. Mol. Biol. Evol. 33, 1337–1348 (2016). PubMed PubMed Central CAS Google Scholar
4.Engel, M. S. The taxonomy of recent and fossil honey bees (Hymenoptera: Apidae; Apis). J. Hymenoptera Res. 8, 165–196 (1999).
Google Scholar
5.Meixner, M. D. et al. Standard methods for characterising subspecies and ecotypes of Apis mellifera. J. Apic. Res. 52, 1–28 (2013). ADS Google Scholar
6.Ruttner, F. Biogeography and Taxonomy of Honeybees (Springer, Berlin, 1988). Google Scholar
7.Yunusbaev, U. B. et al. The role of whole-genome studies in the investigation of honey bee biology. Russ. J. Genet. 55, 815–824 (2019). CAS Google Scholar
8.Bustamante, T., Baiser, B. & Ellis, J. D. Comparing classical and geometric morphometric methods to discriminate between the South African honey bee subspecies Apis mellifera scutellata and Apis mellifera capensis (Hymenoptera: Apidae). Apidologie https://doi.org/10.1007/s13592-019-00651-6 (2019). Article Google Scholar
9.Čermák, K. & Kašpar, F. A method of classifying honey bee races by their body characters. Pszczelnicze Zeszyty Naukowe 44, 81–86 (2000). Google Scholar
10.Bouga, M. et al. A review of methods for discrimination of honey bee populations as applied to European beekeeping. J. Apic. Res. 50, 51–84 (2011). Google Scholar
11.Oleksa, A. & Tofilski, A. Wing geometric morphometrics and microsatellite analysis provide similar discrimination of honey bee subspecies. Apidologie 46, 49–60 (2015). Google Scholar
12.Adam, B. In Search of the Best Strains of Bees. (Walmar Verlag Zell, 1968).
13.De la Rúa, P., Fuchs, S. & Serrano, J. Biogeography of European honey bees. in Beekeeping and Conserving Biodiversity of Honey Bees 15–52 (Northern Bee Books, 2005).
14.Hepburn, H. R. & Crewe, R. M. Portrait of the Cape honeybee, Apis mellifera capensis. Apidologie 22, 567–580 (1991).Google Scholar
15.Uzunov, A. et al. Swarming, defensive and hygienic behaviour in honey bee colonies of different genetic origin in a pan-European experiment. J. Apic. Res. 53, 248–260 (2014). Google Scholar
16.Büchler, R. et al. The influence of genetic origin and its interaction with environmental effects on the survival of Apis mellifera L. colonies in Europe. J. Apic. Res. 53, 205–214 (2014). Google Scholar
17.Francis, R. M. et al. Effect of genotype and environment on parasite and pathogen levels in one apiary—a case study. J. Apic. Res. 53, 230–232 (2014). Google Scholar
18.Requier, F. et al. The conservation of native honey bees is crucial. Trends Ecol. Evol. 34, 789–798 (2019). PubMed Google Scholar
19.Altaye, S. Z., Meng, L. & Li, J. Molecular insights into the enhanced performance of royal jelly secretion by a stock of honeybee (Apis mellifera ligustica) selected for increasing royal jelly production. Apidologie 50, 436–453 (2019). CAS Google Scholar
20.Dogantzis, K. A. & Zayed, A. Recent advances in population and quantitative genomics of honey bees. Curr. Opinion Insect Sci. 31, 93–98 (2019). Google Scholar
21.Ruttner, F., Tassencourt, L. & Louveaux, J. Biometrical-statistical analysis of the geographical variability of Apis mellifera. Apidologie 9, 363–381 (1978). Google Scholar
22.Wilson, E. O. The Insect Societies (Belknap Press, London, 1971). Google Scholar
23.Cridland, J. M., Tsutsui, N. D. & Ramírez, S. R. The complex demographic history and evolutionary origin of the Western honey bee, Apis mellifera. Genome Biol. Evol. 9, 457–472 (2017). PubMed PubMed Central Google Scholar
24.Boardman, L. et al. The complete mitochondrial genome of Apis mellifera unicolor (Insecta: Hymenoptera: Apidae), the Malagasy honey bee. Mitochondrial DNA Part B 4, 3286–3287 (2019). PubMed PubMed Central Google Scholar
25.Boardman, L. et al. The mitochondrial genome of the Carniolan honey bee, Apis mellifera carnica (Insecta: Hymenoptera: Apidae). Mitochondrial DNA Part B 4, 3288–3290 (2019). PubMed PubMed Central Google Scholar
26.Boardman, L. et al. The mitochondrial genome of Apis mellifera simensis (Hymenoptera: Apidae), an Ethiopian honey bee. Mitochondrial DNA Part B 5, 9–10 (2020). Google Scholar
27.Boardman, L. et al. The complete mitochondrial genome of Apis mellifera jemenitica (Insecta: Hymenoptera: Apidae), the Arabian honey bee. Mitochondrial DNA Part B 5, 875–876 (2020). PubMed PubMed Central Google Scholar
28.Boardman, L. et al. The mitochondrial genome of the Maltese honey bee, Apis mellifera ruttneri (Insecta: Hymenoptera: Apidae). Mitochondrial DNA Part B 5, 877–878 (2020). PubMed PubMed Central Google Scholar
29.Boardman, L. et al. The mitochondrial genome of the Spanish honey bee, Apis mellifera iberiensis (Insecta: Hymenoptera: Apidae), from Portugal. Mitochondrial DNA Part B 5, 17–18 (2020). Google Scholar
30.Boardman, L. et al. The complete mitochondrial genome of the West African honey bee Apis mellifera adansonii (Insecta: Hymenoptera: Apidae). Mitochondrial DNA Part B 5, 11–12 (2020). Google Scholar
31.Engel, M. S. Fossil honey bees and evolution in the genus Apis (Hymenoptera: Apidae). Apidologie 29, 265–281 (1998). Google Scholar
32.Kotthoff, U., Wappler, T. & Engel, M. S. Miocene honey bees from the Randeck Maar of southwestern Germany (Hymenoptera, Apidae). Zookeys https://doi.org/10.3897/zookeys.96.752 (2011). Article PubMed PubMed Central Google Scholar
33.Kotthoff, U., Wappler, T. & Engel, M. S. Greater past disparity and diversity hints at ancient migrations of European honey bee lineages into Africa and Asia. J. Biogeogr. 40, 1832–1838 (2013). Google Scholar
34.Nel, A., Martınez-Delclòs, X., Arillo, A. & Peñalver, E. A review of the Eurasian fossil species of the bee Apis. Palaeontol. 42, 243–285 (1999). Google Scholar
35.Culliney, T. W. Origin and evolutionary history of the honeybees Apis. Bee World 64, 29–38 (1983). Google Scholar
36.Deodikar, G. B., Thakar, C. V. & Shah, P. N. Cyto-genetic studies in Indian honey-bees. Proc. Indian Acade. Sci. Sect. B 49, 194–206 (1959). Google Scholar
37.Deodikar, G. B., Thakar, C. V. & Tonapi, K. V. Evolution in the genus Apis. Indian Bee J. 23, 86–91 (1961). Google Scholar
38.Cornuet, J. M. & Garnery, L. Mitochondrial DNA variability in honeybees and its phylogeographic implications. Apidologie 22, 627–642 (1991). CAS Google Scholar
39.Wallberg, A. et al. A worldwide survey of genome sequence variation provides insight into the evolutionary history of the honeybee Apis mellifera. Nat. Genet. 46, 1081–1088 (2014). PubMed CAS Google Scholar
40.Brinkmann, H., van der Giezen, M., Zhou, Y., de Raucourt, G. P. & Philippe, H. An empirical assessment of long-branch attraction artefacts in deep eukaryotic phylogenomics. Syst. Biol. 54, 743–757 (2005). PubMed Google Scholar
41.Franck, P., Garnery, L., Celebrano, G., Solignac, M. & Cornuet, J.-M. Hybrid origins of honeybees from Italy (Apis mellifera ligustica) and Sicily (A. m. sicula). Mol. Ecol. 9, 907–921 (2000). PubMed CAS Google Scholar
42.Franck, P., Garnery, L., Solignac, M. & Cornuet, J.-M. Molecular confirmation of a fourth lineage in honeybees from the Near East. Apidologie 31, 167–180 (2000). CAS Google Scholar
43.Ruttner, F. Naturgeschichte der Honigbienen (Ehrenwirth Verlag, Ehrenwirth, 1992). Google Scholar
44.Franck, P. et al. Genetic diversity of the honeybee in Africa: microsatellite and mitochondrial data. Heredity 86, 420–430 (2001). PubMed CAS Google Scholar
45.Alburaki, M., Moulin, S., Legout, H., Alburaki, A. & Garnery, L. Mitochondrial structure of Eastern honeybee populations from Syria, Lebanon and Iraq. Apidologie 42, 628 (2011). CAS Google Scholar
46.Alburaki, M. et al. A fifth major genetic group among honeybees revealed in Syria. BMC Genet. 14, 117 (2013). PubMed PubMed Central Google Scholar
47.Franck, P., Garnery, L., Solignac, M. & Cornuet, J.-M. The origin of West European subspecies of honeybees (Apis mellifera): New insights from microsatelite and mitochondrial data. Evolution 52, 1119–1134 (1998). PubMed CAS Google Scholar
48.Crozier, R. H. & Crozier, Y. C. The mitochondrial genome of the honeybee Apis mellifera: complete sequence and genome organization. Genetics 133, 97–117 (1993). PubMed PubMed Central CAS Google Scholar
49.Zhang, D. et al. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies. Mol. Ecol. Res. 20, 348–355 (2020). Google Scholar
50.Katoh, K. & Standley, D. M. MAFFT Multiple Sequence Alignment Software Version 7: Improvements in performance and usability. Mol. Biol. Evol. 30, 772–780 (2013). PubMed PubMed Central CAS Google Scholar
51.Rota-Stabelli, O., Lartillot, N., Philippe, H. & Pisani, D. Serine codon-usage bias in deep phylogenomics: Pancrustacean relationships as a case study. Syst. Biol. 62, 121–133 (2013). PubMed CAS Google Scholar
52.Breinholt, J. W. & Kawahara, A. Y. Phylotranscriptomics: Saturated third codon positions radically influence the estimation of trees based on next-gen data. Genome Biol. Evol. 5, 2082–2092 (2013). PubMed PubMed Central CAS Google Scholar
53.Philippe, H. et al. Resolving difficult phylogenetic questions: Why more sequences are not enough. PLoS Biol. 9, e1000602 (2011). PubMed PubMed Central CAS Google Scholar
54.Lartillot, N., Brinkmann, H. & Philippe, H. Suppression of long-branch attraction artefacts in the animal phylogeny using a site-heterogeneous model. BMC Evol. Biol. 7, S4 (2007). PubMed PubMed Central Google Scholar
55.Williams, T. A., Cox, C. J., Foster, P. G., Szöllősi, G. J. & Embley, T. M. Phylogenomics provides robust support for a two-domains tree of life. Nat. Ecol. Evol. 4, 138–147 (2020).PubMed Google Scholar
56.Bleidorn, C. Phylogenomics: An Introduction (Springer International Publishing, Berlin, 2017). https://doi.org/10.1007/978-3-319-54064-1. Book Google Scholar
57.Simion, P. et al. A large and consistent phylogenomic dataset supports sponges as the sister Group to all other animals. Curr. Biol. 27, 958–967 (2017). PubMed CAS Google Scholar
58.Feuda, R. et al. Improved modeling of compositional heterogeneity supports sponges as sister to all other animals. Curr. Biol. 27, 3864-3870.e4 (2017). PubMed CAS Google Scholar
59.Pisani, D. et al. Genomic data do not support comb jellies as the sister group to all other animals. Proc. Natl. Acad. Sci. 112, 15402–15407 (2015). ADS PubMed CAS PubMed Central Google Scholar
60.Betts, H. C. et al. Integrated genomic and fossil evidence illuminates life’s early evolution and eukaryote origin. Nat. Ecol. Evol. 2, 1556–1562 (2018). PubMed PubMed Central Google Scholar
61.Lozano-Fernandez, J. et al. Pancrustacean evolution illuminated by taxon-rich genomic-scale data sets with an expanded remipede sampling. Genome Biol. Evol. 11, 2055–2070 (2019). PubMed PubMed Central Google Scholar
62.Lozano-Fernandez, J. et al. Increasing species sampling in chelicerate genomic-scale datasets provides support for monophyly of Acari and Arachnida. Nat. Commun. 10, 1–8 (2019). Google Scholar
63.Cai, C., Tihelka, E., Pisani, D. & Donoghue, P. C. J. Data curation and modeling of compositional heterogeneity in insect phylogenomics: a case study of the phylogeny of Dytiscoidea (Coleoptera: Adephaga). Mol. Phylogenet. Evol. 147, 106782 (2020). PubMed Google Scholar
64.Lartillot, N., Lepage, T. & Blanquart, S. PhyloBayes 3: A Bayesian software package for phylogenetic reconstruction and molecular dating. Bioinform. 25, 2286–2288 (2009). CAS Google Scholar
65.Lanfear, R., Frandsen, P. B., Wright, A. M., Senfeld, T. & Calcott, B. PartitionFinder 2: New methods for selecting partitioned models of evolution for molecular and morphological phylogenetic analyses. Mol. Biol. Evol. 34, 772–773 (2017). PubMed CAS Google Scholar
66.Nguyen, L.-T., Schmidt, H. A., von Haeseler, A. & Minh, B. Q. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol. Biol. Evol. 32, 268–274 (2015). CAS PubMed Google Scholar
67.Letunic, I. & Bork, P. Interactive tree of life (iTOL) v4: Recent updates and new developments. Nucl. Acids Res. 47, W256–W259 (2019). PubMed CAS PubMed Central Google Scholar
68.Lartillot, N., Rodrigue, N., Stubbs, D. & Richer, J. PhyloBayes MPI: Phylogenetic reconstruction with infinite mixtures of profiles in a parallel environment. Syst. Biol. 62, 611–615 (2013). PubMed CAS Google Scholar
69.Zaitoun, S. T., Al-Ghzawi, A.-M. & Shannag, H. K. Population dynamics of the Syrian Honeybee, Apis mellifera syriaca, under semi-arid Mediterranean conditions. Zool. Middle East 21, 129–132 (2000). Google Scholar
70.Haddad, N. J. et al. Draft genome sequence of the Algerian bee Apis mellifera intermissa. Genom. Data 4, 24–25 (2015). PubMed PubMed Central Google Scholar
71.Arias, M. C. & Sheppard, W. S. Phylogenetic relationships of honey bees (Hymenoptera: Apinae: Apini) inferred from nuclear and mitochondrial DNA sequence data. Mol. Phylogenet. Evol. 37, 25–35 (2005). PubMed CAS Google Scholar
72.Sheppard, W. S. & Meixner, M. D. Apis mellifera pomonella, a new honey bee subspecies from Central Asia. Apidologie 34, 367–375 (2003). Google Scholar
73.Macholán, M. Základy fylogenetické analýzy (Masaryk University, Masaryk, 2014). Google Scholar
74.Garnery, L., Cornuet, J.-M. & Solignac, M. Evolutionary history of the honey bee Apis mellifera inferred from mitochondrial DNA analysis. Mol. Ecol. 1, 145–154 (1992). PubMed CAS Google Scholar
75.Arias, M. C. & Sheppard, W. S. Molecular phylogenetics of honey bee subspecies (Apis mellifera L.) inferred from mitochondrial DNA sequence. Mol. Phylogenet. Evol. 5, 557–566 (1996). PubMed CAS Google Scholar
76.Radloff, S. E. & Hepburn, H. R. Multivariate analysis of honeybee populations, Apis mellifera L (Hymenoptera: Apidae), from western central Africa: morphometrics and pheromones. Afr. Entomol. 5, 195–204 (1997). Google Scholar
77.Amssalu, B., Nuru, A., Radloff, S. E. & Hepburn, H. R. Multivariate morphometric analysis of honeybees (Apis mellifera) in the Ethiopian region. Apidologie 35, 71–81 (2004). Google Scholar
78.Meixner, M. D., Leta, M. A., Koeniger, N. & Fuchs, S. The honey bees of Ethiopia represent a new subspecies of Apis mellifera—Apis mellifera simensis n. ssp. Apidologie 42, 425–437 (2011). Google Scholar
79.Cornuet, J. M. & Fresnaye, J. Etude biométrique de colonies d’abeilles d’Espagne et du Portugal. Apidologie 20, 93–101 (1989). Google Scholar
80.Arias, M. C., Rinderer, T. E. & Sheppard, W. S. Further characterization of honey bees from the Iberian Peninsula by allozyme, morphometric and mtDNA haplotype analyses. J. Apic. Res. 45, 188–196 (2006). CAS Google Scholar
81.Cánovas, F., de la Rúa, P., Serrano, J. & Galián, J. Geographical patterns of mitochondrial DNA variation in Apis mellifera iberiensis (Hymenoptera: Apidae). J. Zool. Syst. Evol. Res. 46, 24–30 (2008). Google Scholar
82.Chávez-Galarza, J. et al. Mitochondrial DNA variation of Apis mellifera iberiensis: further insights from a large-scale study using sequence data of the tRNAleu-cox2 intergenic region. Apidologie 48, 533–544 (2017). Google Scholar
83.Huelsenbeck, J. P. Performance of phylogenetic methods in simulation. Syst. Biol. 44, 17–48 (1995). Google Scholar
84.Felsenstein, J. Inferring Phylogenies (Sinauer Associates, Sunderland, 2004). Google Scholar
85.Kandemir, I., Kence, M., Sheppard, W. S. & Kence, A. Mitochondrial DNA variation in honey bee (Apis mellifera L.) populations from Turkey. J. Apic. Res. 45, 33–38 (2006). CAS Google Scholar
86.Donoghue, P. C. & Benton, M. J. Rocks and clocks: calibrating the Tree of Life using fossils and molecules. Trends Ecol. Evol. 22, 424–431 (2007). PubMed Google Scholar
87.Foord, A. S. Notes on a collection of East Coast amber belonging to Mrs Burwood of Yarmouth. Trans. Norfolk Norwich Nat. Soc. 5, 92–95 (1890). Google Scholar
88.Cockerell, T. D. A. Some European fossil bees. Entomol. 42, 313–317 (1909). Google Scholar
89.Zeuner, F. E. & Manning, F. J. A monograph on fossil bees (Hymenoptera: Apoidea). Bull. Brit. Mus. Nat. Hist. Geol. 27, 151–268 (1976). Google Scholar
90.Stroiński, A. & Szwedo, J. Yuripopoverus africanus gen. et sp. n from East African copal (Hemiptera: Fulgoromorpha: Ricaniidae). Pol. J. Entomol. 80, 679–688 (2011). Google Scholar
91.Ruttner, F., Milner, E. & Dews, J. E. The Dark European Honey Bee, Apis mellifera mellifera Linnaeus 1758. (British Isles Bee Breeders Association, 1990).
92.Bloch, G. et al. Industrial apiculture in the Jordan valley during Biblical times with Anatolian honeybees. Proc. Natl. Acad. Sci. 107, 11240–11244 (2010). ADS PubMed CAS PubMed Central Google Scholar
93.Sheppard, W. S. & Berlocher, S. H. Allozyme variation and differentiation among four Apis species. Apidologie 20, 419–431 (1989). Google Scholar
94.Ramírez, S. R. et al. A molecular phylogeny of the stingless bee genus Melipona (Hymenoptera: Apidae). Mol. Phylogenet. Evol. 56, 519–525 (2010). PubMed Google Scholar
95.Felsenstein, J. Confidence limits on phylogenies: An approach using the bootstrap. Evolution 39, 783–791 (1985). PubMed Google Scholar
96.Young, A. D. & Gillung, J. P. Phylogenomics—principles, opportunities and pitfalls of big-data phylogenetics. Syst. Entomol. n/a, (2019).
97.Eme, L., Spang, A., Lombard, J., Stairs, C. W. & Ettema, T. J. G. Archaea and the origin of eukaryotes. Nat. Rev. Microbiol. 15, 711–723 (2017). PubMed CAS Google Scholar
98.Crewe, R. M., Hepburn, H. R. & Moritz, R. F. A. Morphometric analysis of two southern African races of honeybee. Apidologie 25, 61–70 (1994). Google Scholar
99.Harpur, B. A., Minaei, S., Kent, C. F. & Zayed, A. Management increases genetic diversity of honey bees via admixture. Mol. Ecol. 21, 4414–4421 (2012). PubMed Google Scholar
100.Harpur, B. A., Minaei, S., Kent, C. F. & Zayed, A. Admixture increases diversity in managed honey bees: Reply to De la Rúa et al.. Mol. Ecol. 22, 3211–3215 (2013). PubMed Google Scholar
101.De la Rúa, P., Jaffé, R., DallOlio, R., Muñoz, I. & Serrano, J. Biodiversity, conservation and current threats to European honeybees. Apidologie 40, 263–284 (2009). Google Scholar
102.de la Rúa, P. et al. Conserving genetic diversity in the honeybee: Comments on Harpur et al. (2012). Mol. Ecol. 22, 3208–3210 (2013). PubMed Google Scholar
103.Harpur, B. A. et al. Assessing patterns of admixture and ancestry in Canadian honey bees. Insect. Soc. 62, 479–489 (2015). Google Scholar
104.Chapman, N. C. et al. Hybrid origins of Australian honeybees (Apis mellifera). Apidologie 47, 26–34 (2016).
Добавить комментарий