Аннотация
Таксономия медоносной пчелы A. mellifera содержит много вопросов, связанных со спецификой структуры популяции, особенностями биологии и разрешенностью методов выделения подвидов медоносной пчелы. Между ареалами подвидов существует множество переходных зон, что привело к постепенному изменению характеристик между соседними подвидами. В данной работе приводится современная таксономическая схема медоносной пчелы Apis mellifera. Тридцать три отдельных подвида медоносной пчелы распространены по всей Африке (11 подвидов), Западной Азии и Ближнему Востоку (9 подвидов) и Европе (13 подвидов). Все подвиды медоносной пчелы подразделяются на 5 эволюционных линий: линия A (10 подвидов) и ее подлиния Z (3 подвида), линия M (3 подвида), линия C (10 подвидов), линия O (3 подвида), линия Y (1 подвид), линия C или O (3 подвида).
Введение
Вид медоносной пчелы Apis mellifera Linnaeus, 1758 распространен в широком ареале с различными климатическими условиями и подразделяется на многочисленные подвиды Ruttner, 1988, Sheppard et al., 1997, Engel, 1999). Различные таксономические ревизии описали 20-30 подвидов для A. mellifera (Buttel-Reepen, 1906, Enderlein, 1906, Скориков, 1929a, Скориков, 1929b, Maa, 1953, Ruttner, 1988, Engel, 1999). Таксономия медоносной пчелы A. mellifera противоречива и сохраняет ряд ошибок и вопросов. Некоторые подвиды медоносной пчелы описаны только по морфометрии без каких-либо молекулярных доказательств (Maa, 1953, Ruttner, 1988, Arias and Sheppard, 1996, Franck et al., 2000, Smith, 2002, Kandemir et al., 2011). Подвидовой состав эволюционных линий A (+Z), M, C, O, Y еще не решен и нуждается во всеобъемлющих молекулярных доказательствах (Smith, 1991, Smith et al., 1997; Garnery et al., 1992; Kandemir and Kence, 1995; Alburaki et al., 2011, Alburaki et al., 2013).
Поскольку подвиды медоносной пчелы описываются как географические расы, путаница в их географической локализации и распространении генов наблюдается в Африке (Ruttner, 1992, Hepburn and Radloff, 1998, Dukku, 2016), в Европе (Ruttner, 1988, Cornuet and Garnery, 1991, Smith, 1991, Franck et al., 1998) и на Ближнем Востоке (Franck et al., 2000, Kandemir et al., 2006; Alburaki et al., 2011, Alburaki et al., 2013). Прояснение всех путаниц в таксономии медоносной пчелы Apis mellifera является основной целью данной работы. Здесь мы описали все новые, старые, синонимичные и неправильно написанные таксономические названия медоносной пчелы A. mellifera.
Подходы к определению таксономической принадлежности медоносной пчелы A. mellifera
Методы определения таксономической принадлежности медоносной пчелы A. mellifera на основе морфометрии подразделяются на две группы: 1. измерение частей тела; 2. анализ формы крыльев. Для различения подвидов медоносной пчелы в классической морфометрии используется измерение 36 признаков частей тела (Ruttner, 1988). Другие популярные морфометрические подходы для идентификации таксономической принадлежности колоний медоносных пчел основаны на анализе формы крыльев. Существует три различных подхода, основанных на морфометрии крыльев, для дискриминации подвидов медоносных пчел: классическая морфометрия крыльев (DuPraw, 1965), DAWINO (Discriminant Analysis with Numerical Output) (www.beedol.cz) и геометрическая морфометрия (Bookstein, 1991). Классическая морфометрия крыла фиксирует вариации формы крыла путем вычисления 11 углов между 18 узлами в венах крыла, которые представляют собой подмножество набора из 17 углов. Метод DAWINO состоит из полного набора углов Дюпрау, дополненного 7 линейными измерениями, 5 индексами и одной площадью. В геометрической морфометрии используются координаты точек Дюпрау, называемых ориентирами, которые анализируются с помощью многомерных статистических методов (Miguel et al., 2011).
Аллозимный метод определения таксономической принадлежности медоносных пчел основан на изменчивости изоферментов. У медоносных пчел шесть ферментов полиморфны: малатдегидрогеназа MDH1 (EC 1.1.1.37) (7 аллелей) (Smith and Glenn, 1995); малик-фермент ME (EC 1.1.1.40) (4 аллеля) (Sheppard and Berlocher, 1984); эстераза EST-3 (EC 3.1.1) (8 аллелей) (Ivanova et al., 2010); щелочная фосфатаза ALP (EC 3.1.3.1) (3 аллеля) (Ivanova et al., 2010); фосфоглюкомутаза PGM (EC 5.4.2.2) (5 аллелей) (Ivanova et al., 2010); гексокиназа HK (EC 2.7.1.1) (5 аллелей) (Del Lama et al., 1990).
Методы идентификации таксономической принадлежности медоносных пчел на основе митохондриальной ДНК (мтДНК) основаны на ее нуклеотидном полиморфизме. Существует три основных направления дискриминации медоносных пчел на основе мтДНК: 1. Полиморфизм длины фрагментов рестрикционных ферментов (RFLP) (Smith and Brown, 1988, Smith et al., 1989, Smith, 1991; Garnery et al, 1992) и RFLP фрагментов, амплифицированных с помощью ПЦР (PCR-RFLP) (Hall and Smith, 1991, Arias and Sheppard, 2005, Stevanovic et al., 2010, Ivanova et al., 2010); 2. Полиморфизм продуктов ПЦР со специфическими (Arias and Sheppard, 2005) или случайными праймерами — амплификация случайно амплифицированной полиморфной ДНК (RAPD) (Hunt and Page, 1994, Suazo et al, 1998, Tunca and Kence, 2011); 3. Секвенирование мтДНК с поиском однонуклеотидных полиморфизмов (SNPs) (Arias and Sheppard, 1996, Arias and Sheppard, 2005, Franck et al, 1998, Franck et al., 2000, Franck et al., 2001, Franck et al., 2001, Collet et al., 2006, Shaibi et al., 2009, Pinto et al., 2012, Pinto et al., 2014, Ильясов и др., 2016, Ильясов и др., 2019). Изучение мтДНК основывалось на анализе полного или частичного митохондриального генома или межгенных последовательностей IGS. Наиболее полиморфной IGS является область, расположенная между генами COX1 и COX2 мтДНК (Hall and Smith, 1991, Crozier et al., 1991, Arias and Sheppard, 1996, Jensen et al., 2005, Il’yasov et al., 2007, Ilyasov et al., 2011, Ilyasov et al., 2016, Ilyasov et al., 2019, Cánovas et al., 2008, Muli et al., 2014).
Методы идентификации таксономической принадлежности медоносных пчел, основанные на ядерной ДНК (нДНК), ориентированы на ее нуклеотидный полиморфизм. Полиморфизм нДНК может быть выявлен с помощью трех основных подходов: 1. Полиморфизм продуктов ПЦР со специфическими (Arias and Sheppard, 2005, Il’yasov et al., 2015, Kent et al., 2011, Kent et al., 2012) или случайными RAPD праймерами амплификации (Hunt and Page, 1994, Waldschmidt et al., 2002, Kumar and Khan, 2014); 3. Секвенирование мтДНК с поиском однонуклеотидных полиморфизмов (SNPs) (Chapman et al., 2015, Chapman et al, 2016, Harpur et al., 2014, Harpur et al., 2015, Henriques et al., 2018, Il’yasov et al., 2015, Ilyasov et al., 2016, Pinto et al., 2014, Wallberg et al, 2019, Whitfield et al., 2006); 4. Цельногеномное секвенирование методами NGS (Weinstock et al., 2007, Whitfield et al., 2006, Wallberg et al., 2014, Wallberg et al., 2019). Наиболее полиморфными локусами нДНК являются микросателлитные повторы — короткие тандемные повторы (STR), которые выявляются методом ПЦР со специфическими праймерами (De La Rúa et al., 2001, Estoup et al., 1995, Franck et al., 2000, Franck et al., 2001, Garnery et al., 1998, Il’yasov et al., 2015, Ilyasov et al., 2016, Miguel et al., 2011, Solignac et al., 2003).
Современная таксономическая картина медоносной пчелы A. Mellifera
Таксономия медоносной пчелы Apis mellifera Linnaeus, 1758 (синоним: Apis mellifica Linnaeus, 1761, Apis gregaria Geoffroy, 1762, Apis cerifera Scopoli, 1770, Apis daurica Fischer von Waldheim, 1843) имеет много нерешенных вопросов. Количество подвидов медоносной пчелы и их подразделение на эволюционные линии пока не вполне ясно (Smith, 1991, Smith et al., 1997; Garnery et al., 1992; Kandemir and Kence, 1995; Alburaki et al., 2011, Alburaki et al., 2013).
Подвиды медоносной пчелы A. mellifera были разделены на три линии (A, M, C) сначала с помощью морфометрии (около 20 подвидов) (Ruttner et al., 1978, Moritz et al., 1986, Smith and Brown, 1988, Smith, 1991; Garnery et al., 1992; Estoup et al., 1995), на четыре линии (A, M, C, O) по морфометрии и молекулярным данным позже (около 25 подвидов) (Ruttner, 1988, Smith and Brown, 1988, Cornuet et al., 1988, Lebdigrissa et al., 1991, Cornuet and Garnery, 1991, Smith, 1991, Estoup et al., 1995, Arias and Sheppard, 1996, Franck et al., 1998, Palmer et al., 2000) и пять линий (A (+сублиния Z), M, C, O, Y) по молекулярным данным окончательно (около 30 подвидов) (Franck et al., 2001; Alburaki et al., 2011, Alburaki et al., 2013, Meixner et al., 2013).
Наибольшую проблему в дискриминации подвидов представляет наличие переходных зон между ареалами подвидов, что приводит к градиентным изменениям морфометрических и молекулярных признаков. Переход наблюдается между 8 подвидами (A. m. adansonii — A. m. jemenitica, A. m. sahariensis — A. m. intermissa, A. m. intermissa — A. m. major, A. m. intermissa — A. m. scutellata, A. m. capensis — A. m. unicolor) в Африке (Ruttner, 1992, Hepburn and Radloff, 1998, Dukku, 2016); между 3 подвидами (A. m. intermissa — A. m. iberiensis, A. m. iberiensis — A. m. mellifera) в Европе (Ruttner, 1988, Cornuet and Garnery, 1991, Smith, 1991, Franck et al, 1998); между 4 подвидами (A. m. meda — A. m. anatoliaca, A. m. anatoliaca — A. m. syriaca, A. m. syriaca — A. m. lamarckii) на Ближнем Востоке (Franck et al., 2000, Kandemir et al., 2006; Alburaki et al., 2011, Alburaki et al., 2013).
Подвиды медоносной пчелы могут быть отнесены к разным методам. Подвиды A. m. adansonii, A. m. monticola, A. m. scutellata, A. m. capensis и A. m. unicolor невозможно различить по морфометрии (Ruttner et al., 1978), по мтДНК (Garnery et al., 1995; Sheppard et al., 1996), но могут быть различимы по другой морфометрии (Ruttner, 1988), аллозимам (Meixner et al., 2000), мтДНК (Franck et al., 2001, Kasangaki et al., 2017, Techer et al., 2017). Подвиды A. m. jemenitica, A. m. adansonii невозможно различить по морфометрии (Radloff et al., 1998), но можно различить по другим морфометрическим признакам (Ruttner, 1988, Dukku, 2016), по мтДНК (Franck et al., 2001). Подвиды A. m. carpathica, A. m. carnica, A. m. macedonica нельзя различить по морфометрии (Radloff et al., 1998), но можно различить по другим морфометриям (Foti et al., 1965, Engel, 1999, Cauia et al., 2008, Mărghitas et al., 2008, Teleky et al., 2009), по мтДНК (Mărghitas et al., 2009, Bouga et al., 2011, Coroian et al., 2014, Сыромятников и др., 2018; Ильясов и др., 2018). Подвиды A. m. rodopica, A. m. macedonica невозможно различить по морфометрии (Engel, 1999), по аллозиму (Bouga et al., 2005, Kandemir et al., 2005, Ivanova et al., 2010), но можно различить по другим морфометрическим признакам (Foti et al., 1965, Engel, 1999, Cauia et al., 2008, Mărghitas et al., 2008, Teleky et al., 2009), по мтДНК (Lazarov, 1936, Tzonev, 1960, Velichkov, 1970, Bouga et al., 2011), по микросателлитам (Nikolova, 2011, Uzunov et al., 2014), по мтДНК (Bouga et al., 2005, Martimianakis et al., 2011; Meixner et al., 2013; Radoslavov et al., 2017).
Подвиды медоносной пчелы могут быть отнесены к эволюционным линиям различными методами. Подвиды A. m. syriaca, A. m. anatoliaca, A. m. meda отнесены к линии O по морфометрии (Ruttner, 1988), по мтДНК (Arias and Sheppard, 1996), но отнесены к линии A (сублиния Z) по мтДНК (Arias and Sheppard, 1996, Franck et al., 2000, Bouga et al., 2005; Alburaki et al., 2011, Alburaki et al., 2013). Подвид A. m. caucasia, отнесенный к линии O по морфометрии (Ruttner, 1988, Adl et al., 2007, Meixner et al., 2007, Kandemir et al., 2011), но отнесенный к линии C по мтДНК (Franck et al., 2000; Alburaki et al., 2011, Alburaki et al., 2013), по аллозимам (Smith, 1991; Garnery et al., 1992; Kandemir and Kence, 1995, Иванова и др., 2011), по мтДНК (Cornuet and Garnery, 1991, Koulianos and Crozier, 1997, Smith et al., 1997, Garnery et al., 1998, Palmer et al., 2000, Franck et al., 2000, Smith, 2002, Özdïl et al., 2009, Mărghitas et al., 2009; Ильясов и др., 2018). Подвиды A. m. cypria, A. m. adami отнесены к линии O по морфометрии (Ruttner, 1988), но отнесены к линии C по мтДНК и аллозимам (Bouga et al., 2005). Подвид A. m. lamarckii, отнесенный к линии A по морфометрии (Ruttner, 1988), но отнесенный к линии O по мтДНК (Arias and Sheppard, 1996). Подвид A. m. jemenitica, отнесенный к линии A по морфометрии (Ruttner, 1988), но отнесенный к линии Y по мтДНК (El-Niweiri and Moritz, 2008; Alburaki et al., 2011, Alburaki et al., 2013, Coulibaly et al., 2019).
В текущем обзоре всей доступной литературы говорится о существовании 33 подвидов медоносной пчелы A. mellifera. Эти 33 подвида подразделяются на пять эволюционных линий: линия A — 10 подвидов, линия A (сублиния Z) — 3 подвида, линия M — 3 подвида, линия C — 10 подвидов, линия O — 3 подвида, линия Y — 1 подвид, неопределенная линия C или O — 3 подвида. Географически эти подвиды распределены в трех регионах: Африка населена 11 подвидами, Западная Азия и Ближний Восток населены 9 подвидами, Европа населена 13 подвидами (табл. 1). Пространственное распределение 33 подвидов медоносной пчелы A. mellifera было показано на географической карте (рис. 1).

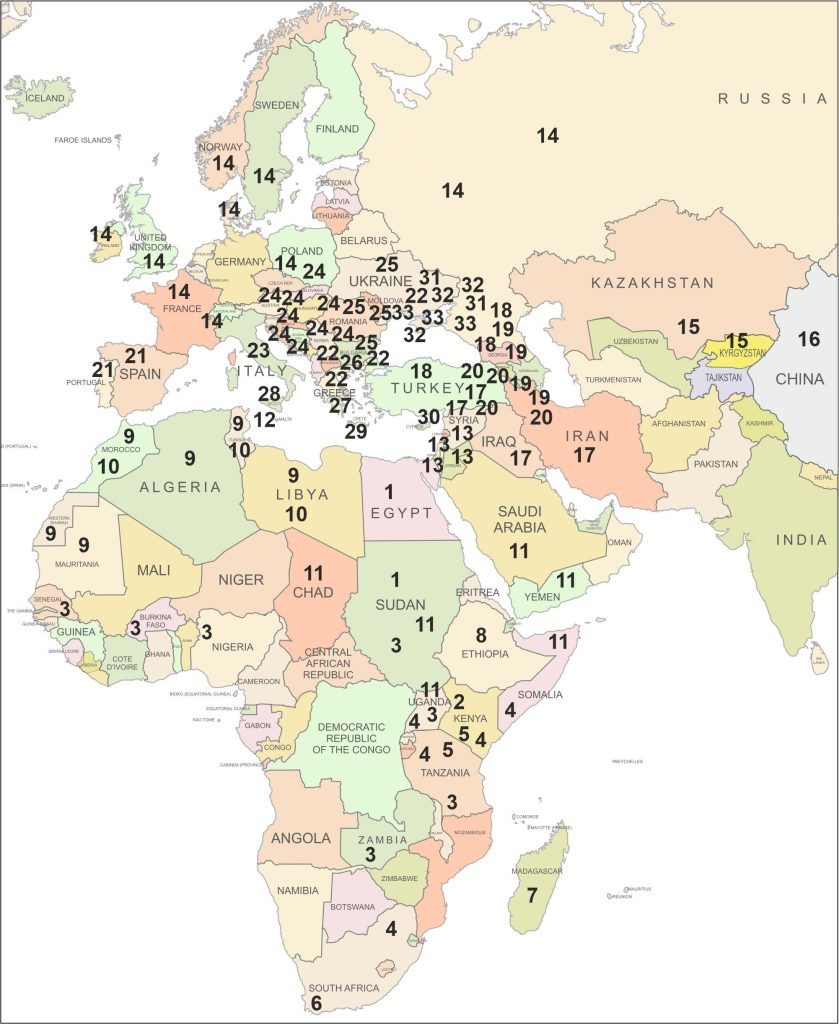
Выводы
Описана современная таксономическая картина медоносной пчелы A. mellifera. Представлены 33 отдельных подвида медоносной пчелы в Африке (11 подвидов), Западной Азии и на Ближнем Востоке (9 подвидов), Европе (13 подвидов), которые подразделяются на 5 эволюционных линий: линия A (10 подвидов), подлиния Z линии A (3 подвида), линия M (3 подвида), линия C (10 подвидов), линия O (3 подвида), линия Y (1 подвид), линия C или O (3 подвида). Таксономия медоносной пчелы A. mellifera имеет много вопросов в связи со спецификой популяционной структуры и разрешенностью методов различения подвидов медоносной пчелы. Переходные зоны между ареалами подвидов медоносной пчелы привели к постепенному изменению всех характеристик между соседями. Между большинством подвидов существует постоянный поток генов из-за отсутствия естественных механизмов изоляции. Некоторые изолированные популяции сохранились на горах и островах, как, например, популяции темных европейских медоносных пчел на Уральских горах (Россия), в Ирландии (Республика Ирландия и Северная Ирландия) и на острове Лезё (Дания). Под руководством профессора Николенко А.Г. по гранту РФФИ № 19-54-70002 e-Asia_t мы изучаем таксономию родственного вида медоносной пчелы Apis cerana на основе молекулярных инструментов, и нашей дальнейшей работой может стать «Пересмотр подвидовой структуры восточной медоносной пчелы Apis cerana».
Ссылки
1.Adl et al., 2007M.B.F. Adl, H.V. Gencer, C. Firatli, R. BahreiniMorphometric characterization of Iranian (Apis mellifera meda), Central Anatolian (Apis mellifera anatoliaca) and Caucasian (Apis mellifera caucasica) honey bee populationsJ. Apic. Res., 46 (4) (2007), pp. 225-231, 10.3896/IBRA.1.46.4.03 View article Google Scholar
2.Alburaki et al., 2011M. Alburaki, S. Moulin, H.E.E. Legout, A. Alburaki, L. GarneryMitochondrial structure of eastern honeybee populations from Syria, Lebanon and IraqApidologie, 42 (5) (2011), pp. 628-641, 10.1007/s13592-011-0062-4
View article View in ScopusGoogle Scholar
3.Alburaki et al., 2013M. Alburaki, B. Bertrand, H. Legout, S. Moulin, A. Alburaki, W.S. Sheppard, et al.A fifth major genetic group among honeybees revealed in SyriaBMC Genet., 14 (2013), pp. 1-10, 10.1186/1471-2156-14-117 View PDF This article is free to access. Google Scholar
4.Arias and Sheppard, 1996M.C. Arias, W.S. SheppardMolecular phylogenetics of honey bee subspecies (Apis mellifera L.) inferred from mitochondrial DNA sequenceMol. Phylogenet. Evol., 5 (3) (1996), pp. 557-566, 10.1006/mpev.1996.0050
View PDF View article View in ScopusGoogle Scholar
5.Arias and Sheppard, 2005M.C. Arias, W.S. SheppardPhylogenetic relationships of honey bees (Hymenoptera: Apinae: Apini) inferred from nuclear and mitochondrial DNA sequence dataMol. Phylogenet. Evol., 37 (1) (2005), pp. 25-35, 10.1016/j.ympev.2005.02.017
View PDF View article View in Scopus Google Scholar
6.Amssalu et al., 2004B. Amssalu, A. Nuru, S.E. Radloff, H.R. HepburnMultivariate morphometric analysis of honeybees (Apis mellifera) in the Ethiopian regionApidologie, 35 (1) (2004), pp. 71-81, 10.1051/apido:2003066 View PDF Google Scholar
7.Bookstein, 1991F.L. BooksteinMorphometric Tools for Landmark Data, Geometry and BiologyCambridge University Press, New York, USA (1991) Google Scholar
8.Bouga et al., 2011M. Bouga, C. Alaux, M. Bienkowska, R. Büchler, N.L. Carreck, E. Cauia, et al.A review of methods for discrimination of honey bee populations as applied to European beekeepingJ. Apic. Res., 50 (2011), pp. 51-84, 10.3896/IBRA.1.50.1.06 View PDF This article is free to access. View in Scopus Google Scholar
9.Bouga et al., 2005M. Bouga, P.C. Harizanis, G. Kilias, S. AlahiotisGenetic divergence and phylogenetic relationships of honey bee Apis mellifera (Hymenoptera: Apidae) populations from Greece and Cyprus using PCR-RFLP analysis of three mtDNA segmentsApidologie, 36 (3) (2005), pp. 335-344, 10.1051/apido:2005021 View PDF View in Scopus Google Scholar
10.Buttel-Reepen, 1906H.V. Buttel-ReepenBeiträge zur systematik, biologie, sowie zur geschichtlichen und geographischen verbreitung der honigbiene (Apis mellifica L.), ihrer varietaten und der iiberigen apis-arten. Mitteilungen aus dem zoologischen museum in BerlinApistica, 3 (1906), pp. 117-201 Google Scholar
11.Cánovas et al., 2008F. Cánovas, J. Serrano, J. Galián, P. De La RúaGeographical patterns of mitochondrial DNA variation in Apis mellifera iberiensis (Hymenoptera: Apidae)J. Zool. Syst. Evol. Res., 46 (1) (2008), pp. 24-30, 10.1111/j.1439-0469.2007.00435.x
View article View in Scopus Google Scholar
12.Cauia et al., 2008E. Cauia, D. Usurelu, L.M. Magdalena, D. Cimponeriu, P. Apostol, A. Siceanu, et al.Preliminary researches regarding the genetic and morphometric characterization of honeybees (A. mellifera L.) from RomaniaSci. Pap. Anim. Sci. Biotechnol., 41 (2) (2008), pp. 278-286 View in Scopus Google Scholar
13.Chapman et al., 2015N.C. Chapman, B.A. Harpur, J. Lim, T.E. Rinderer, M.H. Allsopp, A. Zayed, et al.A SNP test to identify Africanized honeybees via proportion of African ancestryMol. Ecol. Resour., 15 (6) (2015), pp. 1346-1355, 10.1111/1755-0998.12411
View article View in Scopus Google Scholar
14.Chapman et al., 2016N.C. Chapman, B.A. Harpur, J. Lim, T.E. Rinderer, M.H. Allsopp, A. Zayed, et al.Hybrid origins of Australian honeybees (Apis mellifera)Apidologie, 47 (1) (2016), pp. 26-34, 10.1007/s13592-015-0371-0 View PDF This article is free to access.
View in Scopus Google Scholar
15.Collet et al., 2006T. Collet, K.M. Ferreira, M.C. Arias, A.E.E. Soares, M.A. Del LamaGenetic structure of Africanized honey bee populations (Apis mellifera L.) from Brazil and Uruguay viewed through mitochondrial DNA COI-COII patternsHeredity, 97 (2006), pp. 329-335, 10.1038/sj.hdy.6800875 View PDF This article is free to access. View in Scopus Google ScholarCoroian et al., 2014
16.Cornuet et al., 1988J.-M. Cornuet, A. Daoudi, E.H. Mohssine, J. FresnayeEtude biometrique de colonies d’abeilles marocainesApidologie, 19 (1988), pp. 355-366 View PDF CrossRef View in Scopus Google Scholar
17.Cornuet and Garnery, 1991J.-M. Cornuet, L. GarneryMitochondrial DNA variability in honeybees and its phylogeographic implicationsApidologie, 22 (6) (1991), pp. 627-642, 10.1051/apido:19910606 View PDF View in Scopus Google Scholar
18.Coroian et al., 2014C.O. Coroian, I. Munoz, E.A. Schluns, O.R. Paniti-Teleky, S. Erler, E.M. Furdui, et al.Climate rather than geography separates two European honeybee subspeciesMol. Ecol., 23 (9) (2014), pp. 2353-2361, 10.1111/mec.12731
View article View in Scopus Google Scholar
19.Coulibaly et al., 2019Coulibaly, K.a.S., Majeed, M.Z., Chen, C., Yeo, K., Shi, W., Ma, C.S., 2019. Insights into the maternal ancestry of Côte d’Ivoire honeybees using the intergenic region COI-COII. Insects 10 (4). https://doi.org/10.3390/Insects10040090. Google Scholar
20.Crozier et al., 1991Y.C. Crozier, S. Koulianos, R.H. CrozierAn improved test for Africanized honey bee mitochondrial DNAExperientia, 47 (1991), pp. 968-969 View in Scopus Google Scholar
21.De La Rúa et al., 2001P. De La Rúa, J. Galian, J. Serrano, R.F.A. MoritzMolecular characterization and population structure of the honeybees from the Balearic Islands (Spain)Apidologie, 32 (5) (2001), pp. 417-427, 10.1051/apido:2001141
View PDF View in Scopus Google Scholar
22.Del Lama et al., 1990M.A. Del Lama, J.A. Lobo, A.E.E. Soares, S.N. Del LamaGenetic differentiation estimated by isozymic analysis of Africanized honey bee populations from Brazil and from Central AmericaApidologie, 21 (1990), pp. 271-280, 10.1051/apido:19900401
View PDF View in Scopus Google Scholar
23.Dukku, 2016U.H. DukkuEvaluation of morphometric characters of honeybee (Apis mellifera L.) populations in the Lake Chad Basin in Central AfricaAdv. Entomol., 04 (02) (2016), pp. 75-89, 10.7287/peerj.preprints.1680v1
View PDF View in Scopus Google Scholar
24.Dupraw, 1965E.J. DuprawThe recognition and handling of honeybee specimens in non-Linnean taxonomyJ. Apic. Res., 4 (1965), pp. 71-84, 10.1080/00218839.1965.11100107
View article View in Scopus Google Scholar
25.El-Niweiri and Moritz, 2008M.a.A. El-Niweiri, R.F.A. MoritzMitochondrial discrimination of honeybees (Apis mellifera) of SudanApidologie, 39 (5) (2008), pp. 566-573, 10.1051/apido:2008039
View article View in Scopus Google Scholar
26.Enderlein, 1906G. EnderleinNew honeybees and contribution to the distribution of the genus ApisStettiner Entomologische Zeitung, 67 (1906), pp. 331-334 Google Scholar
27.Engel, 1999M.S. EngelThe taxonomy of recent and fossil honey bees (Hymenoptera, Apidae, Apis)J. Hymenoptera Res., 8 (2) (1999), pp. 165-196, 10.1007/978-1-4614-4960-7_18
View article View in Scopus Google Scholar
28.Estoup et al., 1995A. Estoup, L. Garnery, M. Solignac, J.-M. CornuetMicrosatellite variation in honey bee (Apis mellifera L.) populations: hierarchical genetic structure and test of the infinite allele and stepwise mutation modelsGenetics, 140 (1995), pp. 679-695, 10.1080/00218839.1999.11100990
View PDF View in Scopus Google Scholar
29.Foti et al., 1965Foti, N., Lungu, M., Pelimon, C., Barac, I., Copaitici, M., Marza, E., 1965. Researches on morphological characteristics and biological features of the bee population in Romania. In: Proceedings of the Proceedings of XXth Jubiliar International Congress of Beekeeping Apimondia, Bucharest, Romania, pp. 171–176. Google Scholar
30.Franck et al., 2001P. Franck, L. Garnery, A. Loiseau, B.P. Oldroyd, H.R. Hepburn, M. Solignac, et al.Genetic diversity of the honeybee in Africa: microsatellite and mitochondrial dataHeredity, 86 (4) (2001), pp. 420-430, 10.1046/j.1365-2540.2001.00842.x View PDF This article is free to access. View in Scopus Google Scholar
31.Franck et al., 1998P. Franck, L. Garnery, M. Solignac, J.-M. CornuetThe origin of west European subspecies of honeybees (Apis mellifera): new insights from microsatellite and mitochondrial dataEvolution, 52 (4) (1998), pp. 1119-1134, 10.2307/2411242
View article View in Scopus Google Scholar
32.Franck et al., 2000P. Franck, L. Garnery, M. Solignac, J.-M. CornuetMolecular confirmation of a fourth lineage in honeybees from the Near EastApidologie, 31 (2000), pp. 167-180, 10.1051/apido:2000114 View article View in Scopus Google Scholar
33.Garnery et al., 1998L. Garnery, P. Franck, E. Baudry, D. Vautrin, J.M. Cornuet, M. SolignacGenetic diversity of the west European honey bee (Apis mellifera mellifera and A. m. iberica). II. Microsatellite lociGenet. Sel. Evol., 30 (Suppl 1) (1998), pp. 49-74, 10.1186/1297-9686-30-s1-s49 View PDF This article is free to access.
Google Scholar
34.Garnery et al., 1992L. Garnery, J.M. Cornuet, M. SolignacEvolutionary history of the honey bee Apis mellifera inferred from mitochondrial DNA analysisMol. Ecol., 1 (3) (1992), pp. 145-154, 10.1111/j.1365-294X.1992.tb00170.x
View article View in Scopus Google Scholar
35.Garnery et al., 1995L. Garnery, E.H. Mosshine, B.P. Oldroyd, J.-M. CornuetMitochondrial DNA variation in Moroccan and Spanish honey bee populationsMol. Ecol., 4 (1995), pp. 465-472, 10.1111/j.1365-294X.1995.tb00240.x
View article View in Scopus Google Scholar
36.Hall and Smith, 1991H.G. Hall, D.R. SmithDistinguishing African and European honey bee matrilines using amplified mitochondrial DNAPNAS, 88 (1991), pp. 4548-4552, 10.1073/pnas.88.10.4548
View PDF View in Scopus Google Scholar
37.Harpur et al., 2015B.A. Harpur, N.C. Chapman, L. Krimus, P. Maciukiewicz, V. Sandhu, K. Sood, et al.Assessing patterns of admixture and ancestry in Canadian honey beesInsectes Soc., 62 (4) (2015), pp. 479-489, 10.1007/s00040-015-0427-1
View article View in Scopus Google Scholar
38.Harpur et al., 2014B.A. Harpur, C.F. Kent, D. Molodtsova, J.M.D. Lebon, A.S. Alqarni, A.A. Owayss, et al.Population genomics of the honey bee reveals strong signatures of positive selection on worker traitsProc. Natl. Acad. Sci. USA, 111 (7) (2014), pp. 2614-2619, 10.1073/pnas.1315506111
View PDF View in Scopus Google Scholar
39.Hepburn and Radloff, 1998Hepburn, H.R., Radloff, S.E., 1998. Honeybees of Africa Springer Berlin Heidelberg. https://doi.org/10.1007/978-3-662-03604-4. Google Scholar
40.Henriques et al., 2018D. Henriques, A. Wallberg, J. Chavez-Galarza, J.S. Johnston, M.T. Webster, M.A. PintoWhole genome SNP-associated signatures of local adaptation in honeybees of the Iberian PeninsulaSci. Rep., 8 (1) (2018), p. 11145, 10.1038/s41598-018- 29469-5
View article View in Scopus Google Scholar
41.Hunt and Page, 1994G.J. Hunt, R.E.J. PageLinkage analysis of sex determination in the honey bee (Apis mellifera)Mol. Gen. Genet., 244 (5) (1994), pp. 512-518, 10.1007/BF00583902
View article View in Scopus Google Scholar
42.Ilyasov et al., 2011R.A. Ilyasov, I.A. Kutuev, A.V. Petukhov, A.V. Poskryakov, A.G. NikolenkoPhylogenetic relationships of dark European honeybees Apis mellifera mellifera L. from the Russian Ural and West European populationsJ. Apicult. Sci., 55 (1) (2011), pp. 67-76
View in Scopus Google Scholar
43.Ilyasov et al., 2018R.A. Ilyasov, J. Park, J. Takahashi, H.W. KwonPhylogenetic uniqueness of honeybee Apis cerana from the Korean peninsula inferred from the mitochondrial, nuclear, and morphological dataJ. Apicul. Sci., 62 (2) (2018), pp. 189-214, 10.2478/JAS-2018-0018
View article View in Scopus Google Scholar
44.Ilyasov et al., 2019R.A. Ilyasov, A.G. Nikolenko, V.R. Tuktarov, K. Goto, J.I. Takahashi, H.W. KwonComparative analysis of mitochondrial genomes of the honey bee subspecies A. m. caucasica and A. m. carpathica and refinement of their evolutionary lineagesJ. Apic. Res., 58 (4) (2019), pp. 567-579, 10.1080/00218839.2019.1622320
View article View in Scopus Google Scholar
45.Il’yasov et al., 2007R.A. Il’yasov, A.V. Petukhov, A.V. Poskryakov, A.G. NikolenkoLocal honeybee (Apis mellifera mellifera L.) populations in the UralsRussian J. Genet., 43 (6) (2007), pp. 709-711
View in Scopus Google Scholar
46.Il’yasov et al., 2015R.A. Il’yasov, A.V. Poskryakov, A.V. Petukhov, A.G. NikolenkoGenetic differentiation of local populations of the dark European bee Apis mellifera mellifera L. in the UralsRussian J. Genet., 51 (7) (2015), pp. 677-682, 10.1134/S1022795415070042
View article View in Scopus Google Scholar
47.Ilyasov et al., 2016R.A. Ilyasov, A.V. Poskryakov, A.V. Petukhov, A.G. NikolenkoNew approach to the mitotype classification in black honeybee Apis mellifera mellifera and Iberian honeybee Apis mellifera iberiensisRussian J. Genet., 52 (2016), pp. 281-291, 10.1134/S1022795416020058View article View in Scopus Google Scholar
48.Ivanova et al., 2011E. Ivanova, M. Bienkowska, P. PetrovAllozyme polymorphism and phylogenetic relationships in Apis mellifera subspecies selectively reared in Poland and BulgariaFolia Biol., 59 (2011), pp. 3-4, 10.3409/fb59_3-4.121-126
View article Google Scholar
49.Ivanova et al., 2010E. Ivanova, T. Staykova, P. PetrovAllozyme variability in populations of local Bulgarian honey beeBiotechnol. Biotechnol. Equip., 24 (2) (2010), pp. 371-374, 10.1080/13102818.2010.10817868 View PDF This article is free to access. Google Scholar
50.Jensen et al., 2005A.B. Jensen, K.A. Palmer, J.J. Boomsma, B.V. PedersenVarying degrees of Apis mellifera ligustica introgression in protected populations of the black honeybee, Apis mellifera mellifera, in northwest EuropeMol. Ecol., 14 (1) (2005), pp. 93-106, 10.1111/j.1365-294X.2004.02399.x
View article View in Scopus Google Scholar
51.Kandemir and Kence, 1995I. Kandemir, A. KenceAllozyme variability in a central Anatolian honeybee (Apis mellifera L.) populationApidologie, 26 (1995), pp. 503-510, 10.1051/apido:19950607 View PDF View in Scopus Google Scholar
52.Kandemir et al., 2005I. Kandemir, M. Kence, A. KenceMorphometric and electrophoretic variation in different honeybee (Apis mellifera L.) populationsTurkish J. Vet. Anim. Sci., 29 (3) (2005), pp. 885-890View in ScopusGoogle Scholar
53.Kandemir et al., 2006I. Kandemir, M. Kence, W.S. Sheppard, A. KenceMitochondrial DNA variation in honey bee (Apis mellifera L.) populations from TurkeyJ. Apic. Res., 45 (1) (2006), pp. 33-38, 10.1080/00218839.2006.11101310
View article View in Scopus Google Scholar
54.Kandemir et al., 2011I. Kandemir, A. Ozkan, S. FuchsReevaluation of honeybee (Apis mellifera) microtaxonomy: a geometric morphometric approachApidologie, 42 (5) (2011), pp. 618-627, 10.1007/s13592-011-0063-3
View article View in Scopus Google Scholar
55.Kasangaki et al., 2017P. Kasangaki, G. Nyamasyo, P. Ndegwa, R. Kajobe, C. Angiro, A. Kato, et al.Mitochondrial DNA (mtDNA) markers reveal low genetic variation and the presence of two honey bee races in Uganda’s agro-ecological zonesJ. Apic. Res., 56 (2) (2017), pp. 112-121, 10.1080/00218839.2017.1287997
View article View in Scopus Google Scholar
56.Kent et al., 2011C.F. Kent, A. Issa, A.C. Bunting, A. ZayedAdaptive evolution of a key gene affecting queen and worker traits in the honey bee, Apis melliferaMol. Ecol., 20 (24) (2011), pp. 5226-5235, 10.1111/j.1365-294X.2011.05299.x
View article View in Scopus Google Scholar
57.Kent et al., 2012C.F. Kent, S. Minaei, B.A. Harpur, A. ZayedRecombination is associated with the evolution of genome structure and worker behavior in honey beesProc. Natl. Acad. Sci. USA, 109 (44) (2012), pp. 18012-18017, 10.1073/pnas.1208094109
View article View in Scopus Google Scholar
58.Kerr, 1992W.E. KerrAbelhas Africanas su introduction y expansion en el continente Americano; Subspecies y ecotipas AfricanosIndustrial Apiculture, 13 (1992), pp. 12-21View in ScopusGoogle Scholar
59.Koulianos and Crozier, 1997S. Koulianos, R.H. CrozierMitochondrial sequence characterisation of Australian commercial and feral honeybee strains, Apis mellifera L. (Hymenoptera: Apidae), in the context of the species worldwideAust. J. Entomol., 36 (1997), pp. 359-364, 10.1111/j.1440-6055.1997.tb01486.x View PDF This article is free to access.
View in Scopus Google Scholar
60.Kumar and Khan, 2014Y. Kumar, M.S. KhanGenetic variability of European honey bee, Apis mellifera in mid hills, plains and tarai region of IndiaAfr. J. Biotechnol., 13 (8) (2014), pp. 916-925, 10.5897/AJB2013.13142 View PDF Google Scholar
61.Lazarov, 1936A. LazarovBrief contribution for the study of local Bulgarian beeWorks Bulgarian Natural. Soc., 6 (1936), pp. 156-158 Google Scholar
62.Lebdigrissa et al., 1991K. Lebdigrissa, K. Msadda, J.-M. Cornuet, J. FresnayePhylogenetic relationships between the Tunisian honeybee Apis mellifera intermissa and neighbouring African and west Mediterranean honeybee breedsLandbouwtijdschrift, 44 (1991), pp. 1231-1238 Google Scholar
63.Maa, 1953T.C. MaaAn inquiry into the systematics of the tribus Apidini or honeybees (Hymenoptera)Treubia, 21 (1953), pp. 525-640
View in Scopus Google Scholar
64.Mărghitas et al., 2009Mărghitas, L.A., Dezmirean, D., Teleky, O.R., Furdui, E., Moise, A., Stan, L., et al., 2009. Biodiversity testing of Transylvanian honeybee populations using mtDNA markers. Bull. Univ. Agric. Sci. Veterinary Med. Anim. Sci. Biotechnol. 66 (1–2), 402–406. https://doi.org/10.15835/buasvmcn-asb:66:1-2:3391. Google Scholar
65.Mărghitas et al., 2008L.A. Mărghitas, O. Teleky, D. Dezmirean, M. Rodica, C. Bojan, C. Coroian, et al.Morphometric differences between honey bees Apis mellifera carpatica populations from Transylvanian areaLucrări stiintifice Zootehnie si Biotehnologii, 41 (2) (2008), pp. 309-315 View in Scopus Google Scholar
66.Martimianakis et al., 2011S. Martimianakis, E. Klossa-Kilia, M. Bouga, G. KiliasPhylogenetic relationships of Greek Apis mellifera subspecies based on sequencing of mtDNA segments (COI and ND5)J. Apic. Res., 50 (1) (2011), pp. 42-50, 10.3896/IBRA.1.50.1.05View article View in ScopusGoogle Scholar
67.Meixner et al., 2000M.D. Meixner, M.C. Arias, W.S. SheppardMitochondrial DNA polymorphisms in honey bee subspecies from KenyaApidologie, 31 (2000), pp. 181-190 View PDF View in Scopus Google Scholar
68.Meixner et al., 2011M.D. Meixner, M.A. Leta, N. Koeniger, S. FuchsThe honey bees of Ethiopia represent a new subspecies of Apis mellifera — Apis mellifera simensis n. sspApidologie, 42 (3) (2011), pp. 425-437, 10.1007/s13592-011-0007-y
View article View in Scopus Google Scholar
69.Meixner et al., 2013M.D. Meixner, M.A. Pinto, M. Bouga, P. Kryger, E. Ivanova, S. FuchsStandard methods for characterising subspecies and ecotypes of Apis melliferaJ. Apic. Res., 52 (4) (2013), pp. 1-28, 10.3896/IBRA.1.52.4.05 View PDF This article is free to access.
Google Scholar
70.Meixner et al., 2007M.D. Meixner, M. Worobik, J. Wilde, S. Fuchs, N. KoenigerApis mellifera mellifera in eastern Europe — morphometric variation and determination of its range limitsApidologie, 38 (2007), pp. 191-197, 10.1051/apido:2006068
View article View in Scopus Google Scholar
71.Miguel et al., 2011I. Miguel, M. Baylac, M. Iriondo, C. Manzano, L. Garnery, A. EstonbaBoth geometric morphometric and microsatellite data consistently support the differentiation of the Apis mellifera M evolutionary branchApidologie, 42 (2) (2011), pp. 150-161, 10.1051/apido/2010048 View article View in Scopus Google Scholar
72.Moritz et al., 1986R.F.A. Moritz, C.F. Hawkins, R.H. Crozier, A.G. MackinlayA mitochondrial DNA polymorphism in honeybees (Apis mellifera L.)Experientia, 42 (3) (1986), pp. 322-324, 10.1007/BF01942522
View article View in Scopus Google Scholar
73.Muli et al., 2014E. Muli, H. Patch, M. Frazier, J. Frazier, B. Torto, T. Baumgarten, et al.Evaluation of the distribution and impacts of parasites, pathogens, and pesticides on honey bee (Apis mellifera) populations in East AfricaPLoS ONE, 9 (4) (2014), 10.1371/journal.pone.0094459
View PDF Google Scholar
74.Nikolova, 2011S. NikolovaGenetic variability of local Bulgarian honey bees Apis mellifera macedonica (rodopica) based on microsatellite DNA analysisJ. Apicult. Sci., 55 (2) (2011), pp. 117-129
View in Scopus Google Scholar
75.Özdïl et al., 2009F. Özdïl, M.A. Yildiz, H.G. HallMolecular characterization of Turkish honey bee populations (Apis mellifera) inferred from mitochondrial DNA RFLP and sequence resultsApidologie, 40 (5) (2009), pp. 570-576, 10.1051/apido/2009032 View PDF This article is free to access.View in Scopus Google Scholar
76.Palmer et al., 2000M.R. Palmer, D.R. Smith, O. KaftanogluTurkish honeybees: genetic variation and evidence of a fourth lineage of Apis mellifera mtDNAJ. Hered., 91 (2000), pp. 42-46, 10.1093/jhered/91.1.42
View PDF View in Scopus Google Scholar
77.Pinto et al., 2014M.A. Pinto, D. Henriques, J. Chávez-Galarza, P. Kryger, L. Garnery, R. Van Der Zee, et al.Genetic integrity of the dark European honey bee (Apis mellifera mellifera) from protected populations: a genome-wide assessment using SNPs and mtDNA sequence dataJ. Apic. Res., 53 (2014), pp. 269-278, 10.3896/IBRA.1.53.2.08 View PDF This article is free to access.
View in Scopus Google Scholar
78.Pinto et al., 2012M.A. Pinto, I. Munoz, J. Chavez-Galarza, P. De La RúaThe Atlantic side of the Iberian Peninsula: A hot-spot of novel African honey bee maternal diversityApidologie, 43 (2012), pp. 663-673, 10.1007/s13592-012-0141-1
View article View in Scopus Google Scholar
79.Radloff et al., 1998S.E. Radloff, H.R. Hepburn, S. FuchsEcological and morphological differentiation of the honeybees. Apis mellifera Linnaeus (Hymenoptera: Apidae). of West AfricaAfr. Entomol., 6 (1) (1998), pp. 17-23
View in Scopus Google Scholar
80.Radoslavov et al., 2017G. Radoslavov, P. Hristov, R. Shumkova, I. Mitkov, D. Sirakova, M. BougaA specific genetic marker for the discrimination of native Bulgarian honey bees (Apis mellifera rodopica): duplication of COI gene fragmentJ. Apic. Res., 56 (3) (2017), pp. 196-202, 10.1080/00218839.2017.1307713
View article View in Scopus Google Scholar
81.Ruttner, 1988Ruttner, F., 1988. Biogeography and taxonomy of honeybees. Springer-Verlag Berlin Heidelberg GmbH, 288. https://doi.org/10.1016/0169-5347(89)90176-6. Google Scholar
82.Ruttner, 1992Ruttner, F., 1992. Naturgeschichte der Honigbienen. 455. Google Scholar
83.Ruttner et al., 1978F. Ruttner, L. Tassencourt, J. LouveauxBiometrical statistical analysis of the geographic variability of Apis mellifera LApidologie, 9 (1978), pp. 363-382, 10.1051/apido:19780408 View PDF Google Scholar
84.Shaibi et al., 2009T. Shaibi, S. Fuchs, R.F.A. MoritzMorphological study of honeybees (Apis mellifera) from LibyaApidologie, 40 (2009), pp. 97-105, 10.1051/apido/2008068
View article View in Scopus Google Scholar
85.Sheppard et al., 1997W.S. Sheppard, M.C. Arias, A. Grech, M.D. MeixnerApis mellifera ruttneri, a new honey bee subspecies from MaltaApidologie, 28 (1997), pp. 287-293, 10.1051/apido:19970505
View PDF View in Scopus Google Scholar
86.Sheppard and Berlocher, 1984W.S. Sheppard, S.H. BerlocherEnzyme polymorphism in Apis mellifera from NorwayJ. Apic. Res., 23 (1984), pp. 64-69, 10.1080/00218839.1984.11100610
View article View in Scopus Google Scholar
87.Sheppard et al., 1996Sheppard, W.S., Rinderer, T.E., Meixner, M.D., Yoo, H.R., Stelzer, J.A., Al., E., 1996. Hinfl variation in mitochondrial DNA of old world honey bee subspecies. J. Heredity 87, 35–40. https://doi.org/10.1093/oxfordjournals.jhered.a022950. Google Scholar
88.Skorikov, 1929aA.S. SkorikovBeiträge zur Kenntnis der Kenntnis der kaukasischen HonigbienenrassenRep. Bureau Appl. Entomol. (Russia), 4 (1929), pp. 1-59 Google Scholar
89.Skorikov, 1929bA.S. SkorikovEine neue Basis fur eine Revision der Gattung Apis LRep. Bureau Appl. Entomol. (Russia), 4 (1929), pp. 249-270 View in Scopus Google Scholar
90.Smith, 1991D.R. SmithMitochondrial DNA and honeybee biogeographyD.R. Smith (Ed.), Diversity in the genus Apis, Westview Press, Boulder, CO (1991), pp. 131-176 View in Scopus Google Scholar
91.Smith, 2002D.R. SmithGenetic diversity in Turkish honey beesUludağ Arıcılık Dergisi, 2 (2002), pp. 10-17, 10.1080/09064700600641681 View article Google Scholar
92.Smith and Brown, 1988D.R. Smith, W.M. BrownPolymorphisms in mitochondrial DNA of European and Africanized honey bees (Apis mellifera)Experientia, 44 (1988), pp. 257-260, 10.1007/BF01941730
View article Google Scholar
93.Smith et al., 1997D.R. Smith, B.J. Crespi, F.L. BooksteinFluctuating asymmetry in the honey bee, Apis mellifera: effects of ploidy and hybridizationJ. Evol. Biol., 10 (4) (1997), pp. 551-574, 10.1007/s000360050041
View article View in Scopus Google Scholar
94. Smith and Glenn, 1995D.R. Smith, T.C. GlennAllozyme polymorphisms in Spanish honeybees (Apis mellifera iberica)J. Hered., 86 (1) (1995), pp. 12-16, 10.1093/oxfordjournals.jhered.a111518
View article Google Scholar
95.Smith et al., 1989D.R. Smith, O.R. Taylor, W.M. BrownNeotropical Africanized honey bees have African mitochondrial DNANature, 339 (1989), pp. 213-215, 10.1038/339213a0
View article View in Scopus Google Scholar
96.Solignac et al., 2003M. Solignac, D. Vautrin, A. Loiseau, F. Mougel, E. Baudry, A. Estoup, et al.Five hundred and fifty microsatellite markers for the study of the honeybee (Apis mellifera L.) genomeMol. Ecol. Notes, 3 (2003), pp. 307-311, 10.1046/j.1471-8286.2003.00436.x View article View in Scopus Google Scholar
97.Stevanovic et al., 2010J. Stevanovic, Z. Stanimirovic, M. Radakovic, S.R. KovacevicBiogeographic study of the honey bee (Apis mellifera L.) from Serbia, Bosnia and Herzegovina and Republic of Macedonia Based on mitochondrial DNA analysesRussian J. Genet., 46 (5) (2010), pp. 603-609, 10.1134/S1022795410050145
View article View in Scopus Google Scholar
98.Suazo et al., 1998A. Suazo, R. Mctiernan, H.G. HallDifferences between African and European honey bees (Apis mellifera L.) in random amplified polymorphic DNA (RAPD)J. Hered., 89 (1) (1998), pp. 32-36, 10.1093/jhered/89.1.32
View PDF View in Scopus Google Scholar
99.Syromyatnikov et al., 2018M.Y. Syromyatnikov, A.V. Borodachev, A.V. Kokina, V.N. PopovA molecular method for the identification of honey bee subspecies used by beekeepers in RussiaInsects, 9 (2018), p. E10, 10.3390/insects9010010
View PDF Google Scholar
100.Techer et al., 2017M.A. Techer, J. Clémencet, C. Simiand, P. Turpin, L. Garnery, B. Reynaud, et al.Genetic diversity and differentiation among insular honey bee populations in the southwest Indian Ocean likely reflect old geographical isolation and modern introductionsPLoS ONE, 12 (12) (2017), Article e0189234, 10.1371/journal.pone.0189234 View PDF View in Scopus Google Scholar
101.Teleky et al., 2009Teleky, O.R., Bojan, C., Moise, A., Coroian, C., Dezmirean, D., Mărghitaș, L.A., 2009. Ecotypes differentiation within honeybee (Apis mellifera carpatica) from Transylvania. Bulletin of University of Agricultural Sciences and Veterinary Medicine. Anim. Sci. Biotechnol. 64, 1–6. https://doi.org/http://dx.doi.org/10.15835/buasvmcn-asb:64:1-2:2256. Google Scholar
102.Tunca and Kence, 2011R.I. Tunca, M. KenceGenetic diversity of honey bee (Apis mellifera L.: hymenoptera: Apidae) populations in Turkey revealed by RAPD markersAfr. J. Agric. Res., 6 (2011), pp. 6217-6225, 10.5897/AJAR10.386
View article View in Scopus Google Scholar
103.Tzonev, 1960I. TzonevBiometric investigations on the A. mellifera (methods, season dynamics and family changeability)News Student Scient. Soc. (1960), pp. 157-191
View in Scopus Google Scholar
104.Uzunov et al., 2014A. Uzunov, M.D. Meixner, H. Kiprijanovska, S. Andonov, A. Gregorc, E. Ivanova, et al.Genetic structure of apis mellifera macedonica in the Balkan peninsula based on microsatellite DNA polymorphismJ. Apic. Res., 53 (2) (2014), pp. 288-295, 10.3896/IBRA.1.53.2.10
View article View in Scopus Google Scholar
105.Velichkov, 1970V. VelichkovHoney bee races in BulgariaBeekeeping, 10 (1970), pp. 7-11 Google Scholar
106.Waldschmidt et al., 2002A.M. Waldschmidt, P. Marco-Junior, E.G. Barros, L.A. CamposGenetic analysis of Melipona quadrifasciata Lep. (Hymenoptera: Apidae, Meliponinae) with RAPD markersBraz. J. Biol., 62 (4B) (2002), pp. 923-928, 10.1590/s1519-69842002000500022
View PDF View in Scopus Google Scholar
107.Wallberg et al., 2019A. Wallberg, I. Bunikis, O.V. Pettersson, M.-B. Mosbech, A.K. Childers, J.D. Evans, et al.A hybrid de novo genome assembly of the honeybee, Apis mellifera, with chromosome-length scaffoldsBMC Genomics, 20 (1) (2019), p. 275, 10.1186/s12864-019-5642-0 View PDF This article is free to access. View in Scopus Google Scholar
108.Wallberg et al., 2014A. Wallberg, F. Han, G. Wellhagen, B. Dahle, M. Kawata, N. Haddad, et al.A worldwide survey of genome sequence variation provides insight into the evolutionary history of the honeybee Apis melliferaNat. Genet., 46 (10) (2014), pp. 1081-1088, 10.1038/ng.3077 View article View in Scopus Google Scholar
109.Weinstock et al., 2007G.M. Weinstock, G.E. Robinson, R.A. Gibbs, K.C. Worley, J.D. Evans, R. Maleszka, et al.Insights into social insects from the genome of the honeybee Apis melliferaNature, 443 (7114) (2007), pp. 931-949, 10.1038/nature05260
View PDF This article is free to access. Google Scholar
110.Whitfield et al., 2006C.W. Whitfield, Y. Ben-Shahar, C. Brillet, I. Leoncini, D. Crauser, Y. Leconte, et al.Genomic dissection of behavioral maturation in the honey beeProc. Natl. Acad. Sci., 103 (44) (2006), pp. 16068-16075, 10.1073/pnas.0606909103
View article View in Scopus Google Scholar
Добавить комментарий