Аннотация
Справочная информация
Грибки, связанные с насекомыми, представляют собой потенциально богатый источник для открытия новых метаболитов. Однако комплексное понимание грибковых сообществ Apis mellifera ligustica остается неуловимым.
Результаты
Здесь мы исследовали филогенетическое разнообразие и состав сообщества грибов, связанных с медоносными пчелами, используя комбинацию культурно-зависимого и культурно-независимого подходов. Всего из Apis mellifera ligustica, маточного молочка и сотов было выделено и очищено сорок пять грибков, которые принадлежали к четырем классам и одиннадцати различным родам. Кроме того, из грибкового метагенома методом ПЦР было получено 28 последовательностей генов 16S рРНК бактерий. Анализ высокопроизводительного секвенирования показал, что грибковые сообщества были более разнообразными, в общей сложности 62 рода грибов были обнаружены в кишечнике медоносной пчелы независимым от культуры методом, в то время как только 4 рода были выделены независимым от культуры методом. Аналогичным образом, в сотах было обнаружено 247 видов грибов, в то время как культурозависимым методом было выделено только 4 вида. Кроме того, мы оценили антибактериальную и антиоксидантную активность грибковых изолятов. Большинство сырых экстрактов грибов, полученных из супернатанта культивирования, проявили антиоксидантную активность. Только два неочищенных экстракта грибов проявили умеренную активность против Escherichia coli и Staphylococcus aureus. Химический анализ Chaetomium subaffine MFFC22 привел к обнаружению трех известных соединений, включая кохлиодинол (1), эмодин (2), хризофанол (3). Среди них кохлиодинол (1) проявил интенсивную активность по уничтожению радикалов DPPH с 50% ингибирующей концентрацией (IC50) 3,06 мкг/мл, что было сравнимо с положительной аскорбиновой кислотой (IC50 = 2,25 мкг/мл). Соединение 2 проявило слабую ингибирующую активность в отношении Micrococcus tetragenus и S. aureus.
Выводы
Данное исследование позволило получить фундаментальные сведения о сложных взаимодействиях между медоносными пчелами, грибами, бактериальными симбионтами и их влиянии на медоносную пчелу. Кроме того, разнообразие грибов, ассоциированных с медоносной пчелой, имеет большой потенциал для поиска новых видов и антиоксидантов.
Справочная информация
Насекомые — самая многочисленная группа животных на Земле. Названо более миллиона видов насекомых, которые широко распространены в различных местах обитания [1, 2]. Огромное разнообразие насекомых создает большое микробное сообщество, связанное с насекомыми. Некоторые виды микроорганизмов выполняют ценные функции в питании и защите у социальных насекомых, таких как термиты, муравьи и пчелы [3—5]. Стоит отметить, что отношения между насекомыми и связанными с ними грибами привлекают внимание исследователей, например, симбиоз жука и гриба, культивирующего гриб, термитов, выращивающих гриб, и муравьев, выращивающих гриб [6—8]. Кроме того, сообщается, что некоторые бактерии присутствуют в гифах грибов [9, 10]. Стоит рассмотреть возможность дальнейшей оценки многих аспектов ассоциаций между грибами и насекомыми, а также грибами и бактериями.
Насекомо-ассоциированные грибы являются жизненно важными микробными источниками природных биоактивных продуктов. Насекомо-ассоциированные грибы, выделенные из трупов членистоногих, муравьев-листорезов и каменных мух, содержали новые виды микроорганизмов [11—13]. Насекомо-ассоциированные грибы могут повысить приспособленность насекомых к жизни путем производства биологически активных соединений [14]. Кроме того, из грибов, ассоциированных с насекомыми, было обнаружено много новых соединений, которые могут использоваться в качестве ингибиторов иммунитета, антибактериальных агентов и биофунгицидов [15—17]. Однако современные исследования метаболитов, продуцируемых грибами, ассоциированными с насекомыми, все еще не полны.
Как социальное насекомое, медоносная пчела относится к классу Insecta и порядку Hymenoptera в трибе Arthropoda. Это критически важный вид для сельскохозяйственного производства в качестве опылителей [18]. Различные части личинок и взрослых особей медоносной пчелы, их пища и соты содержат множество микроорганизмов, которые играют важную роль в переваривании пищи, опылении и антагонистическом действии против различных патогенов [19]. Особенно грибки, связанные с медоносной пчелой, могут служить материалом для разложения пыльцы или помогать в созревании маточного молочка, а также могут быть источником пищи [20, 21]. Например, личинка бразильской безжалостной пчелы растет, поедая мицелий гриба Monascus внутри расплодной ячейки [22]. Таким образом, медоносные пчелы являются потенциальной моделью взаимодействия грибков-хозяев и сибионтов, которую стоит изучить. Медоносная пчела Apis mellifera ligustica, производящая большое количество маточного молочка, приобретает грибки из своего рациона, окружающей среды или от товарищей [23]. Однако лишь в нескольких сообщениях приводятся грибки, выделенные из образцов, связанных с A. mellifera ligustica [24]. Наши знания о видах, биологической активности и вторичных метаболитах этих ассоциированных грибов до сих пор ограничены. Здесь мы изучили разнообразие грибов из A. mellifera ligustica с помощью культурно-зависимых и культурно-независимых методов и исследовали бактериальные симбионты, биологическую активность и вторичные метаболиты грибов, выделенных из A. mellifera ligustica (личинки, имаго), сот и маточного молочка.
Результаты
Идентификация культивируемых грибов, ассоциированных с медоносной пчелой
В данном исследовании из сот, маточного молочка, личинок и различных частей медоносной пчелы на девяти различных средах было выделено в общей сложности сорок пять грибков (Таблица 1, Рис. 1). Морфологические и микроскопические характеристики десяти репрезентативных штаммов показаны на рис. 2. Среди них семь штаммов были выделены из сотов, тринадцать — из маточного молочка, десять — из личинок, два — из кутикулы медоносной пчелы, семь — из кишечника, три — из головы и три — из гипофарингеальной железы. Анализ последовательности ITS сорока пяти изолятов показал, что грибковые изоляты принадлежали к двум различным филам (Basidiomycota и Ascomycota) и одиннадцати различным родам. Двадцать семь изолятов (60,0%) были распределены в Агарикомицетах в пределах филума Basidiomycota. Остальные восемнадцать изолятов были сгруппированы в три класса [Sordariomycetes (26,7%), Dothideomycetes (11,1%) и Eurotiomycetes (2,2%)] в пределах Ascomycota.
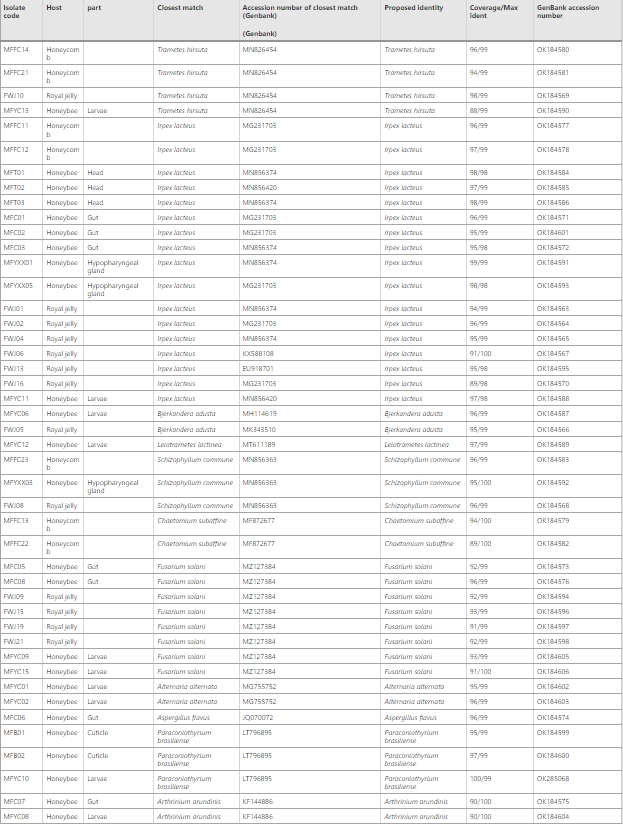


Грибковые изоляты, принадлежащие к классу Agaricomycetes, были отнесены к двум порядкам, включая Agaricales (3 изолята) и Polyporales (24 изолята). Три штамма Agaricales были выделены из сот, маточного молочка и гипофарингеальной железы, соответственно, которые показали высокое сходство с Schizophyllum commune с более чем 99% идентичностью. Три штамма Agaricales были выделены из сот, маточного молочка и гипофарингеальной железы, соответственно, которые показали высокое сходство с Schizophyllum commune с идентичностью более 99%. Штаммы Polyporales были выделены из большинства образцов и представлены семействами Polyporaceae, Irpicaceae и Phanerochaetaceae. Среди них большинство штаммов (17 изолятов), принадлежащих к роду Irpex, были идентифицированы как Irpex lacteus. Примечательно, что штамм FWJ13 показал только 98% сходство с I. lacteus. Кроме того, два штамма, выделенные из личинок медоносной пчелы, были идентифицированы как Bjerkandera adusta и Leiotrametes lactinea, соответственно. Более того, B. adusta также был обнаружен в маточном молочке. Последние четыре штамма, принадлежащие к роду Trametes, показали сходство с Trametes hirsuta с идентичностью 99%.
Другим представительным классом были Sordariomycetes, включающие три порядка (Hypocreales, Sordariales и Xylariales). Восемь изолятов, принадлежащих к семейству Hyporcreaceae, показали совпадение последовательности с Fusarium solani более чем на 99%. Fusarium spp. были обнаружены в ассоциации с кишечником медоносной пчелы, личинками и маточным молочком. Оба штамма, относящиеся к Sordariales, были выделены из сотов и идентифицированы как Chaetomium subaffine с идентичностью 100%. Два других штамма, относящихся к Xylariales, были идентифицированы как Arthrinium arundinis, которые, как было установлено, наиболее связаны с кишечником и личинками медоносной пчелы.
Пять грибковых последовательностей Dothideomycetes были сгруппированы в Pleosporales, включая Paraconiothyrium brasiliense (3 изолята) и Alternaria alternate (2 изолята). P. brasiliense с идентичностью 99% были выделены из кутикулы и личинок медоносной пчелы, соответственно. Род Alternaria был выделен только из личинок медоносной пчелы.
Наконец, только один изолят, принадлежащий к классу Eurotiomycetes, был сгруппирован в порядок Eurotiales, который принадлежал к роду Aspergillus с высоким совпадением последовательности с Aspergillus flavus (> 99%). Штамм был выделен из кишечника медоносной пчелы.
Культурно-независимое сообщество
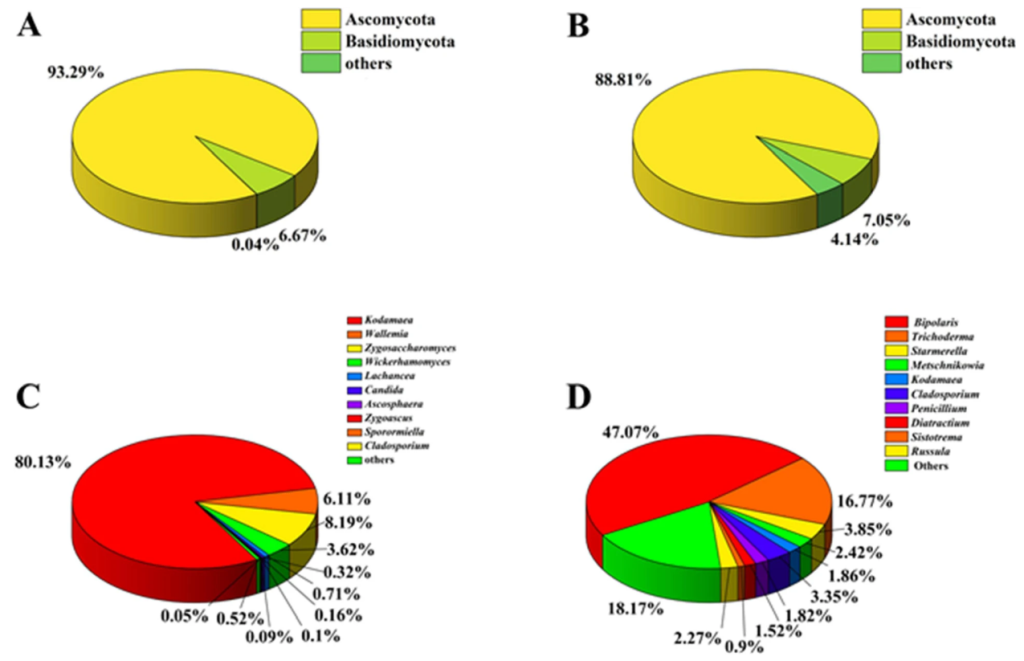
Для анализа грибкового сообщества в кишечнике и сотах медоносной пчелы с помощью секвенирования Illumina Miseq был проведен анализ области ITS1. Всего было получено 408 112 высококачественных чистых грибковых чтений из двух популяций с 3 репликами для каждой популяции (кишечник пчелы: MFF1_1, MFF1_2, MFF1_3, соты: FC_01, FC_02, FC_03) (Дополнительные таблицы S1 и S2). Всего в образцах кишечника медоносной пчелы было идентифицировано 3 грибковых филы, включая Ascomycota, Basidiomycota и неклассифицированные грибы, средняя численность которых составила 93,29, 6,67, 0,04%, соответственно (рис. 3A). В образцах сотов были выявлены Ascomycota (88,81% от средней численности) и Basidiomycota (7,05%) (рис. 3C). Что касается состава грибкового сообщества, во всех образцах на филумном уровне доминировали Ascomycota. Доля Ascomycota в кишечнике пчелы была выше, чем в сотах. Состав сообщества был далее проанализирован на уровне родов. В исследованных образцах кишечника пчелы и сотов было выявлено 62 и 247 родов, соответственно. Среди них Kodamaea (80,13%), Zygosaccharomyces (8,19%), Wallemia (6,11%) и Wickerhamomyces (3,62%) имели более высокую численность в кишечнике пчелы (рис. 3B). Однако Bipolaris (47,07%), Metschnikowia (18,17%), Starmerella (3,85%), Trichoderma (16,77%), Kodamaea (3,35%) имели более высокую численность в сотах (рис. 3D). Относительная численность дрожжей, таких как Kodamaea, Zygosaccharomyces и Wickerhamomyces, была выше в кишечнике пчелы, чем в сотах. Существуют различия в грибковых сообществах образцов кишечника и сотов медоносной пчелы между двумя подходами. Культурно-независимый анализ сообществ показал большее разнообразие грибковых сообществ, чем культурно-зависимый метод.
Идентификация бактериальных симбионтов
В этом исследовании из экстрактов геномной ДНК грибов, ассоциированных с медоносной пчелой, было выделено двадцать восемь бактериальных симбионтов 16S рРНК (Таблица2). Среди них девятнадцать, шесть, два и один штамм были выделены из хозяев Agaricomycetes, Sordariomycetes, Dothideomycetes и Eurotiomycetes, соответственно. Все бактериальные симбионты были разделены на четыре рода путем первоначального BLAST сравнения в GenBank и были предположительно идентифицированы как Bacillus velezensis, Bacillus siamensis, Microbacterium tumbae, Pandoraea sputorum и Achromobacter xylosoxidans. Эти бактериальные симбионты были в основном идентифицированы как B. velezensis и B. siamensis в Bacillaceae. Более того, B. velezensis был получен из грибков различных образцов или частей медоносной пчелы, таких как кишечник, гипофарингеальная железа, голова, кутикула, личинки, соты и маточное молочко.

Антибактериальная активность
Для оценки антибактериальной активности 36 неочищенных экстрактов грибов, полученных из супернатанта культивирования, был использован метод фильтровальной бумаги (Таблица 3). Большинство сырых экстрактов грибов не проявили антибактериальной активности. Однако I. lacteus FWJ16 проявил умеренную активность против E. coli с диаметром зоны ингибирования (ДЗИ) 10,13 мм, что было слабее, чем положительный гентамицина сульфат с ДЗИ 26,33 мм. Кроме того, P. brasiliense MFYC10 также проявил умеренную активность против S. aureus с IZD 9,67 мм, что было слабее, чем положительный гентамицина сульфат с IZD 23,92 мм.

Антиоксидантная активность
Антиоксидантная активность 36 неочищенных экстрактов грибов представлена в таблице 3. Результаты показали, что 32 экстракта (88,9%) проявили антиоксидантную активность при концентрации 166,67 мкг/мл. Среди них, степень поглощения в пределах 10-40%, 40-70% и 70-100% была обнаружена у 19 (52,8%), 3 (8,3%) и 5 (13,9%) штаммов, соответственно. Особенно сырой экстракт MFFC22 показал самую сильную антиоксидантную активность с активностью DPPH-скэвенга 91,6%. Таким образом, MFFC22 был выбран для оценки антиоксидантной активности в различных концентрациях (рис. 4). Результат показал, что MFFC22 обладает более высокой активностью с увеличением концентрации. Его IC50 по активности DPPH scavenging составлял 22,11 мкг/мл, что было относительно слабее, чем коммерческий природный антиоксидант аскорбиновая кислота (Vc, 2,25 мкг/мл).

Идентификация вторичных метаболитов из C. subaffine MFFC22
Три соединения были очищены из жидкого продукта ферментации C. subaffine MFFC22, и их структуры были определены как кохлиодинол (1) [25], эмодин (2) [26] и хризофанол (3) [27] (рис. 5) путем анализа спектроскопических данных и сравнения с литературными данными.

Кохлиодинол (1): фиолетовое твердое вещество; HR-ESI-MS: m/z: 505,2109 [M-H]-, рассчитано для C32H30N2O4 505,2128; 1H ЯМР (600 МГц, CDCl3) δ: 8,42 (1H, s, 2,5-OH), 8,14 (1H, s, 1´-NH), 7,60 (1H, s, H2´), 7. 44 (1H, с, H4´), 7,34 (1H, д 8,3, H7´),7,09 (1H, д 8,3, H6´), 5,40 (1H, м, H11´), 3,48 (1H, д 7,1, H10´), 1,76 (3H, с, 13´-CH3), 1,74 (1H, с, 14´-CH3); 13C ЯМР (150 МГц, CDCl3): 134,3 с (C8´), 133,7 с (C5´), 131,6 с (C12´), 127,1 д (C2´), 126,2 с (C9´), 124,4 д (C11´), 123,4 д (C6´), 120. 8 s (C4´), 111.0 d (C7´), 110.7 s (C3, C6), 104.2 s (C3´), 34.6 t (C10´), 25.6 q (C14´), 17.8 q (C13´). Из-за резонанса бензохинонового кольца, резонансы карбонилов (C1, C2, C4 и C5) кохлиодинола не наблюдались; эти карбонилы можно наблюдать только в THF-d8 или CDCl3 при -75 ℃ [28].
Эмодин (2): оранжевые кристаллы; HR-ESI-MS: m/z: 269.0444 [M-H]-, рассчитано для C15H9O5, 269.0450; 1H ЯМР (600 МГц, ацетон-d6) δ:12.21 (1H, s, 1-OH), 12. 08 (1H, с, 8-OH), 10.15 (1H, с, 3-OH), 7.59 (1H, д 1.5, H5), 7.28 (1H, д 1.5, H4), 7.16 (1H, д 0.6, H7), 6.68 (1H, д 2.0, H2), 2.48 (3H, с, 6-CH3); 13C ЯМР (150 МГц, ацетон-d6): 191.6 (С9), 182.1 (С10), 166.5 (С1), 166.3 (С3), 163.3 (С8), 148.8 (С6), 136.9 (С12), 134.5 (С14), 125.0 (С7), 121.7 (С5), 113.8 (С13), 109.8 (С11), 109.7 (С4), 109.0 (С2), 22.2 (CH3).
Хризофанол (3): оранжевые кристаллы; HR-ESI-MS: m/z: 253,0521 [M-H]-, рассчитано для C15H9O4, 253,0501; 1H ЯМР (600 МГц, CDCl3) δ: 12,12 (1H, с, 1-OH), 12,02 (1H, с, 8-OH), 7,83 (1H, д 7,2, H5), 7,68 (1H, м, H6), 7,66 (1H, с, H4), 7,30 (1H, д 8,4, H7), 7,11(1H, с, H2), 2,47 (3H, с, 3-CH3).
Антибактериальная и антиоксидантная активность соединений
Антибактериальная активность соединений, выделенных из MFFC22, показана в таблице 4. В частности, соединение 2 проявило слабую ингибирующую активность против M. tetragenus и S. aureus с IZD 7,00 и 7,33 мм, соответственно. Соединение 1 не оказывало ингибирующего действия на три бактериальных штамма.
| Соединения | M. tetragenus (мм) | E. coli (мм) | S. aureus (мм) |
| 1 | NI | NI | NI |
| 2 | 7.00 ± 0.00 | NI | 7.33 ± 0.47 |
| Гентамицин сульфатаа | 26.00 ± 0.00 | 20.00 ± 0.00 | 19.00 ± 0.00 |
Антиоксидантная активность соединения 1, выделенного из MFFC22, показана на рис. 4. Примечательно, что 50%-ная ингибирующая концентрация (IC50) кохлиодинола (1) на активность разложения DPPH составила 3,06 мкг/мл, что было сопоставимо с положительной концентрацией аскорбиновой кислоты (2,25 мкг/мл).
Обсуждение
Микробное сообщество среди насекомых очень обширно. Как особый вид микробного сообщества, грибы, связанные с насекомыми, являются источником новых микробных ресурсов [29, 30] и биологически активных природных продуктов [16, 31, 32]. Хотя грибы присутствуют в A. mellifera ligustica, ведущие исследования в основном сосредоточены на сотах, кишечнике пчелы, пыльце и хлебе [23, 33—36]. Насколько нам известно, это первый отчет о разнообразии, антибактериальной и антиоксидантной активности культивируемых грибов из сотов, продукта медоносной пчелы (маточного молочка), личинок и различных частей A. mellifera ligustica. Сорок пять грибков были выделены и охарактеризованы методом разведения и молекулярно-биологической идентификации. Кроме того, три метаболита были очищены и охарактеризованы из C. subaffine MFFC22. Следовательно, грибки, связанные с медоносными пчелами, могут служить источником микробного разнообразия и натуральных продуктов.
Тот же грибок I. lacteus был выделен и идентифицирован из кишечника, головы, гипофарингеальной железы, личинок и маточного молочка медоносной пчелы в нашем исследовании. Это позволяет предположить, что данный грибок широко распространен в A. mellifera ligustica. Некоторые грибки, выделенные из личинок, гипофарингеальных желез, сот и маточного молочка медоносной пчелы, были дереворазрушающими грибками, такими как T. hirsuta, S. commune, B. adusta и L. lactinea [37—39]. Кроме того, род Arthrinium, выделенный из кишечника и личинок медоносной пчелы, был зарегистрирован как патоген растений [40]. По сравнению с грибковым разнообразием других насекомых, некоторые грибки, выделенные из медоносной пчелы, были такими же, как и у других различных насекомых. Например, I. lacteus и P. brasiliense были также выделены из термитов, а F. solani — из жуков [41, 42]. Роды Aspergillus, Alternaria, Chaetomium, Fusarium и Arthrinium уже были обнаружены у медоносной пчелы. Например, Aspergillus, Chaetomium, Fusarium и Alternaria sp. были выделены из кишечника пчелы, а Arthrinium sp. был выделен из пыльцы [23, 33—35]. Однако большинство из этих грибков, представленных в данном исследовании, являются новыми для этой медоносной пчелы. Например, это первое сообщение о выделении T. hirsuta и S. commune из сотов и маточного молочка, I. lacteus из всех частей образцов медоносной пчелы. Кроме того, в кутикуле медоносной пчелы был обнаружен один редкий грибок P. brasiliense, который ранее был обнаружен в кишечнике Acrida cinerea [43].
Всего 62 рода грибов были обнаружены в кишечнике медоносной пчелы независимым от культуры методом. Среди них преобладали роды Kodamaea, Zygosaccharomyces, Wallemia и Wickerhamomyces. Предыдущее исследование, основанное на пиросеквенировании области ITS образцов кишечника медоносной пчелы, полученных из Кореи, показало, что Saccharomyces и Zygosaccharomyces были доминирующими [20]. Однако Starmerella и Hanseniaspora имели высокую относительную численность в образцах кишечника медоносной пчелы, собранных в Италии и Саудовской Аравии [16]. Предыдущие исследования по преобладающим родам не совпадали друг с другом, а также отличались от наших результатов. Предположительно, это связано с разной окружающей средой в местах отбора проб. Некоторые роды, проанализированные с помощью культурально-независимого подхода, были также выделены с помощью культурально-зависимого метода, например, Aspergillus в кишечнике пчелы, Trametes и Chaetomium в сотах. Однако, в целом, культурально-независимые подходы выявили более высокий микробный состав и разнообразие по сравнению с культурально-зависимым методом. Например, в сотах с помощью культурно-независимого метода было обнаружено 247 родов грибов, в то время как изолировано было только 4 рода. Интересно отметить, что Ascosphaera spp., являющаяся возбудителем болезни медоносов у медоносных пчел по литературным данным [44], была обнаружена в кишечнике и сотах медоносной пчелы с помощью культурально-независимого метода, но она еще не была выделена.
Анализ DPPH-радикалов показал, что большинство неочищенных экстрактов грибов, ассоциированных с медоносными пчелами, проявили антиоксидантную активность. Примечательно, что сырой экстракт C. subaffine MFFC22, выделенный из сот, проявил заметную антиоксидантную активность. Уже известно, что представители рода Chaetomium, такие как C. nigricolor, C. globosum, C. cruentum [26, 44, 45], обладают сильной DPPH-спасательной активностью. Тем не менее, C. subaffine и его метаболиты не проявляли активности DPPH. Соединение 1 из MFFC22 продемонстрировало сильную DPPH-спасательную активность со значением IC50 3,06 мкг/мл, сравнимую с положительной аскорбиновой кислотой (Vc). Сравнивая коммерческий антиоксидант BHT с IC50 95,7 мкМ, Dehghan и др. обнаружили, что соединения 2 и 3 также проявили умеренную антиоксидантную активность против DPPH с IC50 271,2 и 297,0 мкМ, соответственно [46]. Фенольные соединения со свободными гидроксильными группами хорошо известны своей сильной антиоксидантной активностью [47]. Поэтому антиоксидантная активность соединений 1-3 может быть обусловлена их фенольными гидроксильными группами. Кроме того, сообщалось, что антиоксидантные соединения, продуцируемые грибами, могут защищать своих хозяев, повышая устойчивость к абиотическим стрессам [48]. Оказывают ли соединения 1-3 аналогичное действие, заслуживает дальнейшего изучения. Кроме того, было показано, что эти выделенные соединения обладают и другими биоактивными свойствами. Например, соединение 1 является общим грибковым метаболитом с цитотоксической активностью против клеточных линий KB, MDA-MB-435 и MRC5 [49]. Соединения 2-3 были производными антрахинона и действовали как противомалярийные и противоаллергические средства [50, 51].
Симбиотические бактерии встречаются у некоторых грибов, что влияет на функции грибов и последующие взаимодействия хозяин-гриб. Однако лишь несколько исследований изучали их влияние в грибах, связанных с насекомыми [52, 53]. Здесь мы нашли первое представление о бактериальных симбионтах в грибах, связанных с медоносной пчелой. Уже было показано, что бактериальный симбионт медоносной пчелы защищает хозяина, вырабатывая ингибирующие соединения или обеспечивая питание [54, 55]. Однако вопрос о том, влияют ли грибково-бактериальные симбионты на рост грибов, ассоциированных с медоносной пчелой, и как они влияют на трехсторонний мутуализм медоносная пчела-гриб-бактерия, еще предстоит изучить.
Выводы
В данном исследовании разнообразие грибов, ассоциированных с медоносными пчелами, было изучено с помощью методов анализа, как зависящих от культуры, так и не зависящих от культуры. Это исследование расширяет наши знания об ассоциированных с медоносной пчелой грибках и еще больше увеличивает количество видов грибков из A. mellifera ligustica. Результаты показывают, что некоторые из этих грибов обладают антибактериальной и антиоксидантной активностью, среди которых наиболее заметным оказался гриб C. subaffine MFFC22. Кроме того, антиоксидантная активность C. subaffine MFFC22 может быть связана с идентифицированными фенольными соединениями. В целом, культивируемые грибы, ассоциированные с медоносной пчелой, дают представление о широко распространенных симбионтах насекомых, которые имеют большой потенциал в поиске ресурсов новых видов и антиоксидантов.
Материалы и методы
Сбор образцов и выделение микроорганизмов
A. mellifera ligustica (включая личинок, взрослых особей), соты и маточное молочко были собраны в Институте пчеловодческих исследований Аньхойского сельскохозяйственного университета, Хэфэй, Китай (GPS: 31◦53ʹN, 117◦20ʹE) в период с апреля по июль 2021 года. Личинки и взрослые особи медоносной пчелы голодали в течение 24 ч, и все образцы хранились при температуре 4 ℃. Сначала семь личинок, семь взрослых особей и один грамм сот помещали отдельно в 10 мл стерильного фосфатно-буферного солевого раствора (PBS) в автоклавированной пробирке объемом 50 мл для получения грибов из внешней изоляции. Затем в ту же пробирку из внешнего помещали 10 мл 75% этанола на 2 мин, с последующим ополаскиванием в стерильном 10 мл 1% отбеливателя с 0,1% твина 20 три раза (по 30 с). Супернатант был удален и заменен 10 мл стерильного раствора PBS. Затем стерильными щипцами вскрывали образцы взрослой медоносной пчелы, чтобы получить голову, кишечник и гипофарингеальную железу. Каждая часть тела была помещена в 10 мл стерильного PBS. Согласно предыдущему отчету, личинки медоносной пчелы, один грамм сот и один грамм маточного молочка были полностью гомогенизированы отдельно в 10 мл стерильного PBS [56]. Затем гомогенаты разбавляли в десятикратной серии (т.е. 10-1, 10-2, 10-3), и аликвоты по 100 мкл из каждого разведения распределяли на девять изолирующих сред (Таблица 5). Чистые колонии грибов из соответствующего разведения переносили на новую среду PDA и инкубировали аэробно при 28 ℃. Все выделенные штаммы грибов сохраняли на средах PDA при 4 ℃ до использования. Грибы были использованы для сохранения в сублимационной сушке с помощью сублимационной сушилки (BTP-3ES; SP Scientific, США) [57] и хранились в нашем институте.

Секвенирование ДНК
Все грибки, ассоциированные с медоносными пчелами, были идентифицированы с помощью молекулярных методов и морфологических характеристик [10, 58, 59]. Каждый грибок культивировали в среде солодового экстракта при температуре 28 ± 0,5 ℃ в течение 7 дней. Затем выделяли геномную ДНК гриба с помощью набора Fast DNA Extraction Kit (Aidlab Biotechnologies Co., Ltd., Пекин, Китай) в соответствии со спецификацией производителя. Праймеры ITS1/ITS4 были использованы для амплификации ITS на основе геномной ДНК гриба. Кроме того, 16S рРНК амплифицировали с помощью праймеров 27F/1492R методом ПЦР из метагенома грибов [10, 60]. Качество продуктов ПЦР визуализировали на 1% агарозном геле методом электрофореза. Каждый продукт был успешно амплифицирован из ПЦР для секвенирования (Tsingke Biotechnology Co., Ltd., Пекин, Китай).
Идентификация грибов и бактериальных симбионтов
Как упоминалось ранее [42], принадлежность всех достигнутых последовательностей, полученных от Tsingke Biotechnology Company, была распознана по имеющимся данным в BLAST из базы данных Национального центра биотехнологической информации (NCBI). Выравнивание последовательностей и филогенетический анализ Neighbor-joining проводили с помощью программы MEGA версии 5.0. Для оценки информации о соединении соседей на основе 1 000 реплик был использован Bootstrap-анализ построения дерева по прочности последовательностей [61]. Полученные ITS-последовательности были депонированы в базе данных GenBank под номерами доступа OK184563-OK184606 и OK285068. Номерами доступа последовательностей 16S рРНК бактерий были OK147622-OK147645 и OK169608-OK169611.
Анализ независимого от культуры сообщества
Предварительная обработка образцов была такой же, как и для получения кишечника и сот медоносной пчелы. Затем из образцов выделяли тотальную геномную ДНК с помощью набора Fast DNA Extraction Kit, концентрацию и чистоту ДНК подтверждали на 2% агарозном геле. Каждый образец проводили в трех экземплярах. Гены ITS1 регионов амплифицировали с использованием специфического праймера (ITS5-1737F и ITS2-2043R) со штрих-кодом. Условия реакции составляли 98℃ в течение 1 мин, затем 30 циклов 98℃ в течение 10 с, 50℃ в течение 30 с и 72℃ в течение 30 с, наконец, 72℃ в течение 5 мин. Продукты ПЦР смеси были очищены с помощью набора Qiagen Gel Extraction Kit (Qiagen, Германия). Библиотеку секвенирования генерировали с помощью набора TruSeq® DNA PCR-Free Sample Preparation Kit (Illumina, США) в соответствии с рекомендациями производителя и добавляли индексные коды. Качество библиотеки оценивали на флуорометре Qubit@ 2.0 (Thermo Scientific) и системе Agilent Bioanalyzer 2100. Наконец, библиотека была секвенирована на платформе Illumina NovaSeq с использованием парных чтений длиной 250 п.н.
Сырые чтения были демультиплексированы и подвергнуты качественной фильтрации с помощью QIIME V1.9.1 [62]. Эффективные метки были получены путем сравнения с эталонной базой данных Unite и использования алгоритма UCHIME для удаления химерных последовательностей [63, 64]. Последовательности с ≥ 97% сходством были отнесены к одним и тем же операционным таксономическим единицам (OTU) с помощью программного обеспечения Uparse V7.0.1001 [65]. Таксономию каждой репрезентативной последовательности оценивали с помощью программы MUSCLE V3.8.31 и сравнивали с базой данных Unite на основе алгоритма blast [66]. Исходные данные доступны в NCBI Short Read Archive под номерами доступа PRJNA817087 и PRJNA817099.
Культивирование грибов и приготовление экстракта культуры
Каждый гриб инокулировали в среду PDA и инкубировали при 28 ± 0,5 ℃ в течение 3-4 дней. Затем свежие мицелии каждого гриба (9 пробок размером 5 мм) инокулировали в 250 мл колбу Эрленмейера, содержащую 150 мл жидкой среды ME, и инкубировали при 28 ± 0,5 ℃ в течение 7 дней в шейкере, вращающемся со скоростью 180 об/мин. Культуру пропускали через четыре слоя марли для удаления таллома гриба, затем надосадочную жидкость трижды экстрагировали этилацетатом (EtOAc, 1:1, v/v). Неочищенные экстракты грибов концентрировали в вакууме, чтобы получить экстракт культуры для дальнейшего экспериментального использования.
Антимикробная активность
Для проверки антибактериальной активности неочищенных экстрактов использовали метод фильтровальной бумаги [67]. Три бактериальных штамма (Escherichia coli (ATCC8739), Micrococcus tetragenus (ATCC35098) и Staphylococcus aureus (ATCC6538) были отобраны для теста и культивированы на триптическом соевом кровяном агаре (TSBA) при 37 ℃. Затем на стерильные диски фильтровальной бумаги (диаметром 5 мм) добавляли по 5 мкл исследуемых неочищенных экстрактов, которые отдельно растворяли в ацетоне для получения концентрации 18 мг/мл. Диски фильтровальной бумаги, обработанные только ацетоном и сульфатом гентамицина, служили отрицательным и положительным контролем, соответственно. Пластины готовили в трех экземплярах и культивировали при 37 ℃ в течение 24-36 ч. Наконец, для оценки антимикробной активности измеряли диаметр зоны ингибирования (в мм).
Антиоксидантная активность
Активность 2,2-дифенил-1-пикрилгидразила (DPPH) по поглощению радикалов проводили в соответствии с предыдущим методом с некоторыми модификациями [68]. 1 мл каждого неочищенного экстракта (1 мг/мл) смешивали с 5 мл раствора DPPH в метаноле (20 мкг/мл). Смесь инкубировали при комнатной температуре в течение 30 мин в темноте, и абсорбцию измеряли при 517 нм с помощью спектрофотометра (UV-1601; Beijing Beifen-ruili Analytical Instrument Co., Ltd., Китай). Метанол и аскорбиновая кислота (Vc) использовались в качестве отрицательного и положительного контроля, соответственно. Каждый образец проводили в трех экземплярах. Радикальная активность сырых экстрактов в DPPH рассчитывалась следующим образом:
Scavenging rate (SR) (%) = (Ab— As) /Ab*100%
где SR — поглощающая активность исследуемого образца (%), Ab — абсорбция без образца, As — абсорбция в присутствии образцов или положительного вещества.
Изоляция соединений из MFFC22
13 л культурального бульона MFFC22 фильтровали и экстрагировали EtOAc (3 × 13 л). Фазу EtOAc концентрировали при пониженном давлении для получения неочищенного экстракта (2 г). Неочищенный экстракт был подвергнут колоночной хроматографии (КХ) с использованием силикагеля (100-200 меш) с градиентом CH2Cl2/MeOH (100:0-100:16, v/v) для получения шести фракций (Fr1-Fr6). Соединение 1 (15 мг) было кристаллизовано из раствора CH2Cl2 из Fr1 (CH2Cl2 /MeOH, 100:0, v/v). Fr1 далее фракционировали на колонке с силикагелем, элюируя с (CH2Cl2/MeOH, 100:0, 100:1, v/v), чтобы получить соединения 2 (0,9 мг) и 3 (0,7 мг).
Структурная элюция метаболитов
Структуры всех соединений были проанализированы в первую очередь с помощью спектрометрии высокого разрешения (HR-ESI-MS) и спектроскопии 1H/13C-ядерного магнитного резонанса (ЯМР). Спектры HR-ESI-MS измерялись на масс-анализаторе TripeTOF 4600 (Bruker, США). Данные ЯМР 1H/13C были получены на спектрометре Agilent DD2 600 Гц (Agilent, США) с тетраметилсиланом (ТМС) в качестве внутреннего стандарта, химические сдвиги (δ) были представлены в виде частей на миллион (ppm).
Антибактериальная и антиоксидантная активность соединений
Исходные данные, подтверждающие выводы данной рукописи, будут предоставлены авторами без излишних оговорок любому квалифицированному исследователю. В данном исследовании были проанализированы общедоступные наборы данных. Эти данные можно найти здесь: Полученные последовательности генов ITS были депонированы в базе данных GenBank под номерами доступа OK184563-OK184606 и OK285068. Полученные последовательности гена 16S рРНК были депонированы в базе данных GenBank под номерами доступа OK147622-OK147645 и OK169608-OK169611. Данные высокопроизводительного секвенирования по технологии paired-end Illumina ампликонов гена ITS 1 могут быть получены из архива коротких отпечатков NCBI под номерами доступа PRJNA817087 и PRJNA817099.
Доступность данных и материалов
Исходные данные, подтверждающие выводы данной рукописи, будут предоставлены авторами без необоснованных оговорок любому квалифицированному исследователю. В данном исследовании были проанализированы общедоступные наборы данных. Эти данные можно найти здесь: Полученные последовательности генов ITS были депонированы в базе данных GenBank под номерами доступа OK184563-OK184606 и OK285068. Полученные последовательности гена 16S рРНК были депонированы в базе данных GenBank под номерами доступа OK147622-OK147645 и OK169608-OK169611. Данные высокопроизводительного секвенирования по технологии paired-end Illumina ампликонов гена ITS 1 могут быть получены из архива коротких отпечатков NCBI под номерами доступа PRJNA817087 и PRJNA817099.
Ссылки
1.Basset Y, Cizek L, Cuénoud P, Didham RK, Guilhaumon F, Missa O, et al. Arthropod diversity in a tropical forest. Science. 2012;388:1481.
Article Google Scholar
2.Stork NE. How many species of insects and other terrestrial arthropods are there on earth? Annu Rev Entomol. 2018;63:31–45.
Article CAS Google Scholar
3.Zilber-Rosenberg I, Rosenberg E. Role of microorganisms in the evolution of animals and plants: the hologenome theory of evolution. Fems Microbiol Rev. 2008;32:723–35.
Article CAS Google Scholar
4.Warnecke F, Luginbühl P, Ivanova N, Ghassemian M, Richardson TH, Stege JT, et al. Metagenomic and functional analysis of hindgut microbiota of a wood-feeding higher termite. Nature. 2007;450:560–5.
Article CAS Google Scholar
5.Kwong WK, Engel P, Koch H, Moran NA. Genomics and host specialization of honey bee and bumble bee gut symbionts. Proc Natl Acad Sci USA. 2014;111:11509–14.
Article CAS Google Scholar
6.Hulcr J, Stelinski LL. The ambrosia symbiosis: from evolutionary ecology to practical management. Annu Rev Entomol. 2017;62:285–303.
Article CAS Google Scholar
7.Li HJ, Yelle DJ, Li C, Yang MY, Ke J, Zhang RJ, et al. Lignocellulose pretreatment in a fungus-cultivating termite. Proc Natl Acad Sci USA. 2017;114:4709–14.
Article CAS Google Scholar
8.Li HJ, Sosa-Calvo J, Horn HA, Pupo MT, Clardy J, Rabeling C, et al. Convergent evolution of complex structures for ant-bacterial defensive symbiosis in fungus-farming ants. Proc Natl Acad Sci USA. 2018;115:10720–5.
Article CAS Google Scholar
9.Hoffman MT, Arnold AE. Diverse bacteria inhabit living hyphae of phylogenetically diverse fungal endophytes. Appl Environ Microb. 2010;76:4063–75.
Article CAS Google Scholar
10.Shao MW, Lu YH, Miao S, Zhang Y, Chen TT, Zhang YL. Diversity, bacterial symbionts and antibacterial potential of gut-associated fungiisolated from the Pantala flavescens Larvae in China. PLoS ONE. 2015;10: e0134542.
Article Google Scholar
11.Attili-Angelis D, Duarte APM, Pagnocca FC, Nagamoto NS, de Vries M, Stielow JB, et al. Novel Phialophora species from leaf-cutting ants (tribe Attini). Fungal Divers. 2014;65:65–75.
Article Google Scholar
12.Jaber S, Mercier A, Knio K, Brun S, Kambris Z. Isolation of fungi from dead arthropods and identification of a new mosquito natural pathogen. Parasite Vector. 2016;9:491.
Article Google Scholar
13.White MM, Valle LG, Lichtwardt RW, Lichtwardt RW, Siri A, Strongman DB, et al. New species and emendations of Orphella: taxonomic and phylogenetic reassessment of the genus to establish the Orphellales, for stonefly gut fungi with a twist. Mycologia. 2018;110:147–78.
Article Google Scholar
14.Kandasamy D, Gershenzon J, Andersson MN, Hammerbacher A. Volatile organic compounds influence the interaction of the Eurasian spruce bark beetle ( Ips typographus) with its fungal symbionts. ISME J. 2019;13:1788–800.
Article CAS Google Scholar
15.Zhang YL, Ge HM, Zhao W, Dong H, Xu Q, Li SH, Li J, et al. Unprecedented immunosuppressive polyketides from Daldinia eschscholzii, a mantis-associated fungus. Angew Chem Int Edit. 2008;47:5823–6.
Article CAS Google Scholar
16.Zhang YL, Zhang J, Jiang N, Lu YH, Wang L, Xu SH, et al. Immunosuppressive polyketides from the mantis-associated Daldinia eschscholzii. J Am Chem Soc. 2011;133:5931–40.
Article CAS Google Scholar
17.Zhang SX, Sun FF, Liu LJ, Bao LY, Fang W, Yin CP, et al. Dragonfly-associated Trichoderma harzianum QTYC77 is not only a potential biological control agent of Fusarium oxysporum f. sp. cucumerinum but also a source of new antibacterial agents. J Agric Food Chem. 2020;68:14161–7.
Article CAS Google Scholar
18.Potts SG, Imperatriz-Fonseca V, Ngo HT, Aizen MA, Biesmeijer JC, Breeze TD, et al. Safeguarding pollinators and their values to human well-being. Nature. 2016;540:220–9.
Article CAS Google Scholar
19.Khan KA, Al-Ghamdi AA, Ghramh HA, Ansari MJ, Ali H, Alamri SA, et al. Structural diversity and functional variability of gut microbial communities associated with honey bees. Microb Pathogenesis. 2019;138: 103793.
Article Google Scholar
20.Yun JH, Jung MJ, Kim PS, Bae JW. Social status shapes the bacterial and fungal gut communities of the honey bee. Sci Rep. 2019;2018:8.
Google Scholar
21.de Paula GT, Menezes C, Pupo MT, Rosa CA. Stingless bees and microbial interactions. Curr Opin Insect Sci. 2021;44:41–7.
Article Google Scholar
22.Menezes C, Vollet-Neto A, Marsaioli AJ, Zampieri D, Fontoura IC, Luchessi AD, et al. A Brazilian social bee must cultivate fungus to survive. Curr Biol. 2015;25:1–5.
Article Google Scholar
23.Callegari M, Crotti E, Fusi M, Marasco R, Gonella E, Noni ID, et al. Compartmentalization of bacterial and fungal microbiomes in the gut of adult honeybees. NPJ Biofilms Microbi. 2021;7:42.
Article Google Scholar
24.Kwong WK, Medina LA, Koch H, Sing KW, Soh EJY, Ascher JS, et al. Dynamic microbiome evolution in social bees. Sci Adv. 2017;3: e1600513.
Article Google Scholar
25.Kingsland SR, Barrow RA. Identification of Chaetoviridin E from a cultured microfungus, Chaetomium sp. and structural reassignment of Chaetoviridins B and D. Aust J Chem. 2009;62:269–74.
Article CAS Google Scholar
26.Mari SH, Varras PC, Atia-tul Wahab, Choudhary IM, Siskos MG, Gerothanassis IP. Solvent-dependent structures of natural products based on the combined use of DFT calculations and 1H-NMR chemical shifts. Molecules. 2019;24:2290.
Article Google Scholar
27.Liu RM, Li AF, Sun AL. Preparative isolation and purification of hydroxyanthraquinones and cinnamic acid from the chinese medicinal herb rheum officinale baill. by high-speed counter-current chromatography. J Chromatogr A. 2004;1052:217–21.
Article CAS Google Scholar
28.Xu GB, He G, Bai HH, Yang T, Zhang GL, Wu LW, et al. Indole alkaloids from Chaetomium globosum. J Nat Prod. 2015;78:1479–85.
Article CAS Google Scholar
29.Pažoutová S, Follert S, Bitzer J, Keck M, Surup F, Šrůtka P, et al. A new endophytic insect-associated daldinia species, recognised from a comparison of secondary metabolite profiles and molecular phylogeny. Fungal Divers. 2013;60:107–23.
Article Google Scholar
30.Nagam V, Aluru R, Shoaib M, Dong GR, Li Z, Pallaval VB, et al. Diversity of fungal isolates from fungus-growing termite Macrotermes barneyi and characterization of bioactive compound from Xylaria escharoidea. Insect Sci. 2021;28:392–402.
Article CAS Google Scholar
31.Ren FX, Zhu SM, Wang B, Li L, Liu XZ, Su RB. Hypocriols A-F, heterodimeric botryane ethers from hypocrea sp., an insect-associated fungus. J Nat Prod. 2016;79:1848–56.
Article CAS Google Scholar
32.Tong ZW, Xie XH, Wang TT, Lu M, Jiao RH, Ge HM. Acautalides A-C, neuroprotective diels−alder adducts from solid-state cultivated acaulium sp. H-JQSF Org Lett. 2021;23:5587–91.
Article CAS Google Scholar
33.Gilliam M, Prest DB. Fungi isolated from the intestinal contents of foraging worker honey bees. Apis mellifera J Invertebr Pathol. 1972;20:101–3.
Article Google Scholar
34.Moubasher AH, Abdel-Sater MA, Soliman Z. Yeasts and filamentous fungi inhabiting guts of three insect species in Assiut. Egypt Mycosphere. 2017;8:1297–316.
Article Google Scholar
35.Zhao YZ, Zhang ZF, Cai L, Peng WJ, Liu F. Four new filamentous fungal species from newly-collected and hive-stored bee pollen. Mycosphere. 2018;9:1089–116.
Article Google Scholar
36.Disayathanoowat T, Li HY, Supapimon N, Suwannarach N, Lumyong S, Chantawannakul P, et al. Different dynamics of bacterial and fungal communities in hive-stored bee bread and their possible roles: a case study from two commercial honey bees in China. Microorganisms. 2020;8:264.
Article CAS Google Scholar
37.Lien NTH, Hieu NV, Hong LT, Anh FT, Thao PTH. Identification of white rot fungus CP9 and its potential application in biopulping. J Biotechnol. 2016;14:721–6.
Google Scholar
38.Jović J, Buntić A, Radovanović N, Petrović B, Mojović L. Lignin-degrading abilities of novel autochthonous fungal isolates Trametes hirsuta F13 and Stereum gausapatum F28. Food Technol Biotechnol. 2018;56:354–65.
Article Google Scholar
39.Horisawa S, Inoue A, Yamanaka Y. Direct ethanol production from lignocellulosic materials by mixed culture of wood rot fungi Schizophyllum commune, Bjerkandera adusta, and Fomitopsis palustris. Fermentation. 2019;5:21.
Article CAS Google Scholar
40.Crous PW, Groenewald JZ. A phylogenetic re-evaluation of Arthrinium. Ima Fungus. 2013;4:133–54.
Article Google Scholar
41.Masuya H, Kajimura H, Tomisawa N, Yamaoka Y. Fungi associated with scolytogenes birosimensis (coleoptera: curculionidae) infesting pittosporum tobira. Environ Entomol. 2012;41:255–64.
Article Google Scholar
42.Xu X, Shao MW, Yin CP, Mao ZC, Shi ZC, Yu XY, et al. Diversity, bacterial symbionts, and antimicrobial potential of termite-associated fungi. Front Microbiol. 2020;11:300.
Article Google Scholar
43.Liu CX, Wang L, Chen JF, Guo ZY, Tu X, Deng ZS, et al. Paraconfuranones A-H, eight new furanone analogs from the insect-associated fungus Paraconiothyrium brasiliense MZ-1. Magn Reson Chem. 2015;53:317–22.
Article CAS Google Scholar
44.Dhayanithy G, Subban K, Chelliah J. Diversity and biological activities of endophytic fungi associated with Catharanthus roseus. BMC Microbiol. 2019;19:22.
Article Google Scholar
45.Zhao SH, Wu XL, Duan XY, Zhou CX, Zhao ZQ, Chen H, et al. Optimal extraction, purification and antioxidant activity of total flavonoids from endophytic fungi of Conyza blinii H. Lév PeerJ. 2021;9: e11223.
Article Google Scholar
46.Dehghan H, Salehi P, Amiri MS. Bioassay-guided purification of α-amylase, α-glucosidase inhibitors and DPPH radical scavengers from roots of rheum turkestanicum. Ind Crop Prod. 2018;117:303–9.
Article CAS Google Scholar
47.Campos JF, de Castro DTH, Damião MJ, Torquato HFV, Paredes-Gamero EJ, Carollo CA, et al. The chemical profile of senna velutina leaves and their antioxidant and cytotoxic effects. Oxid Med Cell Longev. 2016;2016:8405957.
Article Google Scholar
48.Jia M, Chen L, Xin HL, Zheng CJ, Rahman K, Han T, Qin P. A friendly relationship between endophytic fungi and medicinal plants: a systematic review. Front Microbiol. 2016;7:906.
Article Google Scholar
49.Casella TM, Eparvier V, Mandavid H, Bendelac A, Odonne G, Dayan L, et al. Antimicrobial and cytotoxic secondary metabolites from tropical leaf endophytes: Isolation of antibacterial agent pyrrocidine C from Lewia infectoria SNB-GTC2402. Phytochemistry. 2013;96:370–7.
Article CAS Google Scholar
50.Stompor-Gorący M. The health benefits of emodin, a natural anthraquinone derived from rhubarb—a summary update. Int J Mol Sci. 2021;22:9522.
Article Google Scholar
51.Kim SJ, Kim MC, Lee BJ, Park DH, Hong SH, Um JY. Anti-inflammatory activity of chrysophanol through the suppression on NF-kB/caspase-1 activation in vitro and in vivo. Molecules. 2010;15:6436–51.
Article CAS Google Scholar
52.Oh DC, Poulsen M, Currie CR, Clardy J. Dentigerumycin: a bacterial mediator of an ant-fungus symbiosis. Nat Chem Biol. 2009;5:391–3.
Article CAS Google Scholar
53.Wyche TP, Ruzzini AC, Schwab L, Currie CR, Clardy J. Tryptorubin A: A polycyclic peptide from a fungus-derived Streptomycet. J Am Chem Soc. 2017;139:12899–902.
Article CAS Google Scholar
54.Martinson VG, Danforth BN, Minckley RL, Rueppell O, Tingek S, Moran N. A simple and distinctive microbiota associated with honey bees and bumble bees. Mol Ecol. 2011;20:619–28.
Article Google Scholar
55.Miller DL, Smith EA, Newton IL. G. A bacterial symbiont protects honey bees from fungal disease. mBio. 2021;12:e00503-21.
Article CAS Google Scholar
56.Chevrette MG, Carlson CM, Ortega HE, Thomas C, Ananiev GE, Barns KJ, et al. The antimicrobial potential of Streptomyces from insect microbiomes. Nat Commun. 2019;10:516.
Article CAS Google Scholar
57.Ryan MJ, Smith D. Cryopreservation and freeze-drying of fungi employing centrifugal and shelf freeze-drying. Methods Mol Biol. 2007;368:127–40.
Article CAS Google Scholar
58.Li J, Zhong M, Lei XL, Xiao SL, Li AY. Diversity and antibacterial activities of culturable fungi associated with coral Porites Pukoensis. World J Microb Biot. 2014;30:2551–8.
Article CAS Google Scholar
59.Liu S, Ahmed S, Zhang CG, Liu TS, Shao CL, Fang YW. Diversity and antimicrobial activity of culturable fungi associated with sea anemone Anthopleura xanthogrammica. Electron J Biotechn. 2020;44:41–6.
Article CAS Google Scholar
60.José RH, Claudia LR, Antonio VN, Alejandro SA, Roberto RM, Holjes SL, et al. A novel intracellular nitrogen-fixing symbiosis made by Ustilago maydis and Bacillus spp. New Phytol. 2015;207:769–77.
Article Google Scholar
61.Felsenstein J. Confidence limits on phylogenies: an approach using thebootstrap. Evolution. 1985;39:783–91.
Google Scholar
62.Caporaso JG, Kuczynski J, Stombaugh J, Bittingeret K, Bushman FD, Costello EK, et al. QIIME allows analysis of high-throughput community sequencing data. Nat Methods. 2010;7:335–6.
Article CAS Google Scholar
63.Edgar RC, Haas BJ, Clemente JC, Quince C, Knight R. UCHIME improves sensitivity and speed of chimera detection. Bioinformatics. 2011;27:2194–200.
Article CAS Google Scholar
64.Haas BJ, Gevers D, Earl AM, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons. Genome Res. 2011;21:494–504.
Article CAS Google Scholar
65.Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nat Methods. 2013;10:996–8.
Article CAS Google Scholar
66.Edgar RC. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 2004;32:1792–7.
Article CAS Google Scholar
67.Li S, Shao MW, Lu YH, Kong LC, Jiang DH, Zhang YL. Phytotoxic and antibacterial metabolites from Fusarium proliferatum ZS07 isolated from the gut of long-horned grasshoppers. J Agric Food Chem. 2014;62:8997–9001.
Article CAS Google Scholar
68.Zhang YL, Yin CP, Kong LC, Jiang DH. Extraction optimisation, purification and major antioxidant component of red pigments extracted from Camellia japonica. Food Chem. 2011;129:660–4.
Article CAS Google Scholar
Дополнительная информация
Дополнительный файл 1: Таблица S1.
Таксономическое распределение полученных в исследовании OTU и их относительное обилие в каждом кишечнике медоносной пчелы.
Дополнительный файл 2: Таблица S2.
Таксономическое распределение полученных в исследовании OTU и их относительное обилие на каждом соте.
Добавить комментарий