Аннотация
Медоносные пчелы являются одновременно важными опылителями и модельными насекомыми благодаря своей высокоразвитой социальности и управлению колонией. Для лучшего понимания молекулярных механизмов, лежащих в основе управления колонией медоносных пчел, важно исследовать экспрессию генов, предположительно участвующих в физиологии колонии. Хотя количественная ПЦР в реальном времени (qRT-PCR) может использоваться для количественной оценки относительной экспрессии целевых генов, сначала необходимо определить внутренние референсные гены (которые стабильно экспрессируются в различных условиях), чтобы обеспечить точную нормализацию целевых генов. Поэтому для определения надежных референсных генов в колониях медоносной пчелы (Apis mellifera) мы оценили семь генов-кандидатов (ACT, EIF, EF1, RPN2, RPS5, RPS18 и GAPDH) в образцах, собранных из трех типов тканей медоносной пчелы (голова, грудь и брюшко) во все четыре сезона с помощью трех программ анализа (NormFinder, BestKeeper и geNorm). Затем мы проверили различные методы нормализации с использованием каждого из семи эталонных генов и комбинации нескольких генов путем расчета экспрессии каталазы (CAT). Несмотря на то, что гены, отнесенные к наиболее стабильным, немного отличались в зависимости от условий и методов анализа, наши результаты показывают, что RPS5, RPS18 и GAPDH представляют собой оптимальные эталонные гены медоносной пчелы для нормализации целевых генов при анализе qRT-PCR различных образцов тканей медоносной пчелы, собранных в разные сезоны.
Введение
Западная медоносная пчела, Apis mellifera L., играет важную роль в качестве опылителя (1). Кроме того, медоносная пчела считается ключевым модельным насекомым из-за ее относительно сложного поведения, включая социальность, разделение труда и управление колонией (2). Предыдущие исследования показали, что состояние эндокринной системы и экспрессия генов являются важными факторами для гибкого управления колонией медоносной пчелы, которое включает в себя сезонное регулирование разделения труда и динамики популяции (3,4,5). Чтобы расширить наше понимание молекулярных механизмов, лежащих в основе регуляции физиологии колоний медоносных пчел, информация о физиологических функциях генов, предположительно связанных с управлением колониями, может быть определена путем анализа профилей их экспрессии в различные сезоны и в тканях медоносных пчел (6,7).
В количественной ПЦР в реальном времени (qRT-PCR) количественно определяют мРНК (или кДНК) конкретного гена; этот метод широко используется благодаря его относительной скорости, чувствительности, воспроизводимости и точности (8,9). Поэтому qRT-PCR был бы идеальным методом для анализа экспрессии генов медоносной пчелы, предположительно участвующих в пластичности молекулярной физиологии колонии, в образцах, собранных в разные сезоны и в разных тканях. Однако, поскольку результаты qRT-PCR очень чувствительны к исходному количеству РНК в реакции амплификации, интерпретация уровней экспрессии целевых генов в различных условиях приведет к значительным ошибкам без использования надежного внутреннего стандарта (7,8,9,10). Поэтому перед анализом уровней экспрессии целевых генов в разных условиях для точной нормализации данных необходимы референсные гены, чтобы компенсировать различия в количестве РНК в разных образцах медоносных пчел; эти внутренние референсные гены должны показывать сходные уровни транскриптов в разных условиях (8,9,11,12).
Учитывая важность точной нормализации в анализах qRT-PCR, референсные гены были определены и проверены у различных видов насекомых (8,13,14). Согласно предыдущим исследованиям медоносной пчелы, широко используемые эталонные гены были валидированы на различных стадиях развития (6), в мозге после бактериального вызова (15), в различных возрастах и социальных ролях (7,10,16). В частности, в нашем предыдущем исследовании (7) было проведено сравнение сезонной стабильности экспрессии эталонных генов-кандидатов между фуражирами и кормилицами. Тем не менее, эталонные гены еще предстоит сравнить между различными тканями медоносной пчелы, собранными в течение всех четырех сезонов. Поэтому в настоящем исследовании мы стремились определить наиболее надежные эталонные гены среди типов тканей медоносной пчелы и в разные сезоны. В частности, мы собирали рабочих особей в течение четырех сезонов (т.е. весной, летом, осенью и зимой) и готовили образцы РНК из трех тканей (головы, грудной клетки и брюшка). Затем мы выбрали пять генов-кандидатов, которые широко используются в качестве референсных генов для нормализации целевых генов в анализах qRT-PCR (6,17,18,19): β-актин (ACT), фактор инициации эукариотической трансляции (EIF), фактор элонгации 1 (EF1), 26S протеасомный неАТФазный регулятор (RPN2) и 40S рибосомный белок S5 (RPS5). Кроме того, в настоящее исследование были добавлены два гена, 40S рибосомный белок S18 (RPS18) и глицеральдегид-3-фосфат дегидрогеназа (GAPDH), которые ранее были определены как оптимальные референсные гены в голове медоносной пчелы7. Впоследствии мы использовали три программы анализа (NormFinder, BestKeeper и geNorm) для оценки стабильности экспрессии семи кандидатных эталонных генов. Кроме того, семь эталонных генов и комбинация нескольких эталонных генов были проверены путем нормализации экспрессии каталазы (CAT).
Результаты
Специфичность и эффективность амплификации
Перед проведением qRT-PCR были исследованы специфичность и эффективность амплификации. Все продукты ПЦР, амплифицированные с каждым набором праймеров, показали одну полосу в 1% агарозном геле и один акулий пик, обнаруженный на кривой плавления при RT-PCR. Более того, учитывая, что прямые и обратные праймеры для EF1, RPS18 и CAT были разработаны на основе двух разных экзонов, содержащих интрон, наличие одной полосы на агарозном геле и одного пика в RT-PCR для EF1, RPS18 и CAT свидетельствовало об отсутствии загрязнения геномной ДНК (Дополнительный рис. S1). При анализе эффективности ПЦР все семь генов-кандидатов имели коэффициенты линейной регрессии (R2) > 0,997 и эффективность амплификации 92-109% (Таблица 1).

Распределение Cq эталонных генов
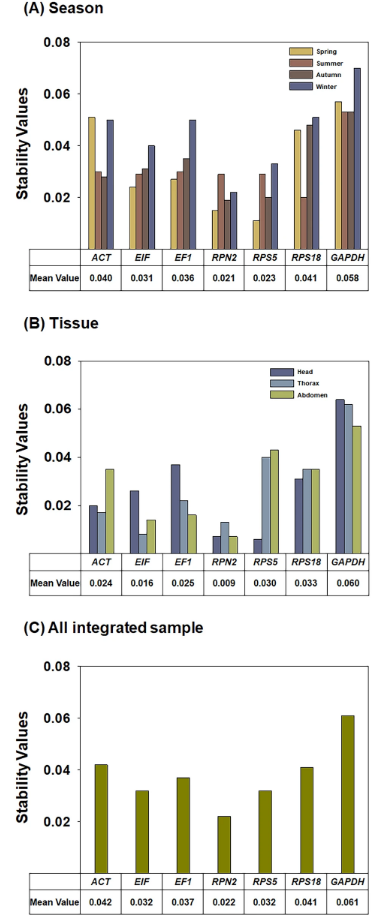
Были проанализированы уровни экспрессии, как показано значениями Cq, семи эталонных генов-кандидатов в образцах транскриптов медоносной пчелы, полученных в течение четырех сезонов и трех типов тканей (рис. 1). На основе арифметических средних (AM) и стандартных отклонений (SD) были рассчитаны значения коэффициента вариации (CV) следующим образом: CV = SD/AM. В течение четырех сезонов EIF демонстрировал наименьшую изменчивость с коэффициентом вариации (CV) 0,02-0,03 (рис. 1A-D). Среди трех типов тканей значения CV для RPN2 были самыми низкими из семи генов (0,01 в голове, 0,03 в грудной клетке и 0,02 в брюшной полости) (рис. 1E-G). EIF также был самым стабильным геном в голове и грудной клетке (рис. 1E,F). Как показано на рис. 1H, при сравнении значений Cq семи генов, полученных из всех образцов в различные сезоны и типы тканей, EIF и RPS18 были наименее изменчивыми генами (CV = 0,03).

Анализ стабильности экспрессии с помощью трех программ
Анализ NormFinder
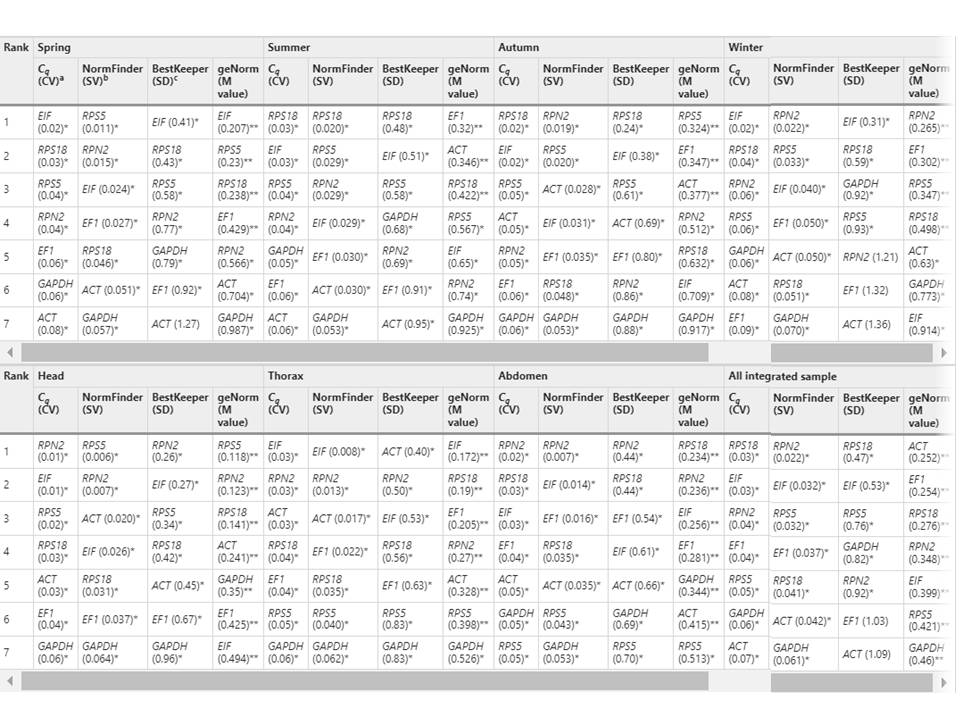
На основе вариаций экспрессии генов-кандидатов программа NormFinder определяет оптимальный референсный ген путем расчета значений стабильности (20,21). Согласно средним значениям стабильности (средние значения), которые были рассчитаны арифметически для четырех сезонов, RPN2 был самым стабильным геном (среднее значение = 0,021) (рис. 2А). При сравнении значений стабильности всех семи генов, RPS5 был самым стабильным геном весной. RPS18 был самым стабильным геном летом, тогда как RPN2 был самым стабильным геном осенью и зимой (рис. 2А, табл. 2). При анализе стабильности генов трех типов тканей, RPN2 был самым стабильным (средняя стабильность = 0,009) (рис. 2B). При анализе стабильности конкретных типов тканей, RPS5 был самым высокоранговым геном в голове (наименее стабильным), в то время как наиболее стабильными уровнями экспрессии в грудной клетке и брюшной полости были EIF и RPN2, соответственно (рис. 2B, табл. 2). Когда значения стабильности генов были рассчитаны путем объединения всех четырех сезонов и трех типов тканей, ранг стабильности от наиболее (наименьшее значение) до наименее (наибольшее значение) был следующим: RPN2 > EIF = RPS5 > EF1 > RPS18 > ACT > GAPDH (рис. 2C, табл. 2).
Анализ BestKeeper
Гены, которые показывают низкие значения SD (обычно < 1) и CV, могут быть выбраны в качестве более стабильных эталонных генов в алгоритме BestKeeper (22,23). Основываясь на значениях SD и CV, BestKeeper выделил EIF (весной и зимой) и RPS18 (летом и осенью) как наиболее подходящие референсные гены с наименьшей вариацией Cq (Таблица 3). Среди трех типов тканей, согласно показателям SD и CV, RPN2 занял первое место в голове и брюшной полости (наименее стабильный ген), в то время как ACT был определен как оптимальный эталонный ген в грудной клетке (Таблица 3). Хотя стабильность генов в голове, грудной клетке и брюшке была различной, все семь генов имели SD < 1,0 во всех тканях, что позволяет предположить, что любой из генов может быть использован в качестве референсного гена для нормализации экспрессии целевых генов в голове, грудной клетке или брюшке медоносных пчел (Таблица 3). Когда значения Cq семи генов были объединены по сезонам и типам тканей, BestKeeper показал, что RPS18, EIF, RPS5, GAPDH и RPN2 имели значения SD < 1,0; таким образом, эти гены, возможно, являются лучшими кандидатами в качестве эталонных генов (Таблица 3, см. Все интегрированные образцы).

Анализ geNorm
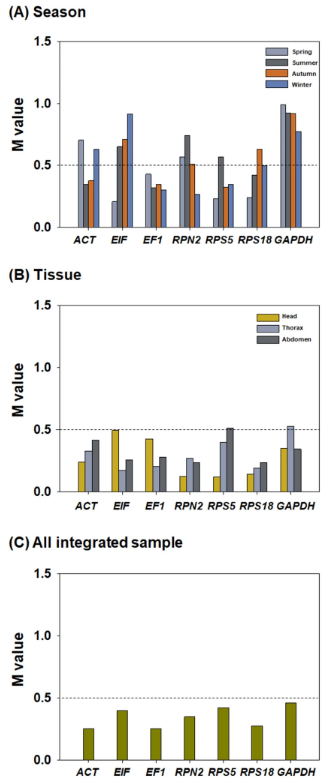
Средние значения стабильности экспрессии (M) семи кандидатов также были определены с помощью geNorm для различных сезонов и типов тканей (рис. 3). M ≤ 0,5 было предложено в качестве критерия для выбора подходящего референсного гена (21,24). При сезонном сравнении значения M для EF1 были < 0,5 в каждом из сезонов, в то время как у остальных шести генов M ≥ 0,5 по крайней мере в одном сезоне (рис. 3A); следовательно, EF1, возможно, является наиболее подходящим эталонным геном для нормализации целевых генов при анализе сезонных тенденций экспрессии генов у медоносных пчел. При сравнении показателей M кандидатов в референсные гены по типам тканей, все гены имели M ≤ 0,5, за исключением GAPDH и RPS5 в грудке и брюшке, соответственно (рис. 3B); следовательно, ACT, EIF, EF1, RPN2 и RPS18 могут быть полезны в качестве референсных генов для анализа экспрессии генов в различных типах тканей медоносной пчелы. Когда значения Cq семи генов, полученных в разные сезоны и из разных типов тканей, были объединены, значения M всех генов были < 0,5 (рис. 3C), что говорит о том, что любой из семи генов может быть эталонным геном согласно анализу geNorm.
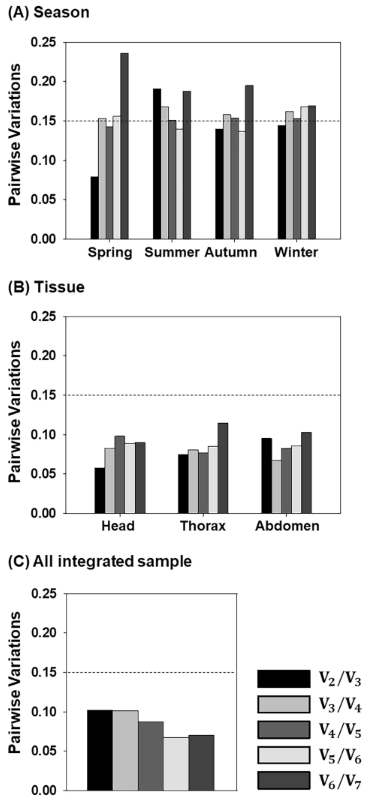
В дополнительном анализе geNorm использовался для расчета значений парной вариации (Vn/Vn+1), которые указывали на оптимальное количество референсных генов для нормализации целевых генов. Согласно предыдущим исследованиям, 0,15 является подходящим значением отсечки при анализе парной вариации (25). При сезонном анализе значения V2/V3 для весенних, осенних и зимних пчел составили 0,079, 0,14 и 0,144, соответственно, что указывает на то, что комбинация двух генов (EIF + RPS5 для весны; RPS5 + EF1 для осени; RPN2 + EF1 для зимы) будет достаточной для нормализации целевых генов в эти сезоны (рис. 4A), исходя из рангов стабильности экспрессии генов, проанализированных с помощью geNorm (показано на рис. 3A, табл. 2). В противоположность этому, только V5/V6 летом был ниже значения отсечки 0,15 (V5/V6 = 0,14) (рис. 4A), что означает, что для нормализации экспрессии целевых генов у летней пчелы необходимо объединить не менее пяти референсных генов. Аналогично, анализ парных вариаций показал, что значения V2/V3 были ниже значения отсечки в голове, грудной клетке и брюшке, что также предполагает, что двух генов (RPS5 + RPN2 для головы; EIF + RPS18 для грудной клетки; и RPS18 + RPN2 для брюшка) будет достаточно для расчета экспрессии генов (рис. 4B). Когда все образцы были объединены, анализ парных вариаций показал, что значения V2/V3 были выше, чем значения V3/V4, V4/V5, V5/V6 и V6/V7; однако V2/V3 составляло 0,102, что все еще было ниже значения отсечки 0,15. Этот результат подтверждает вывод о том, что два гена, ACT и EF1, были оптимальными факторами нормализации для анализа экспрессии генов (рис. 4C).
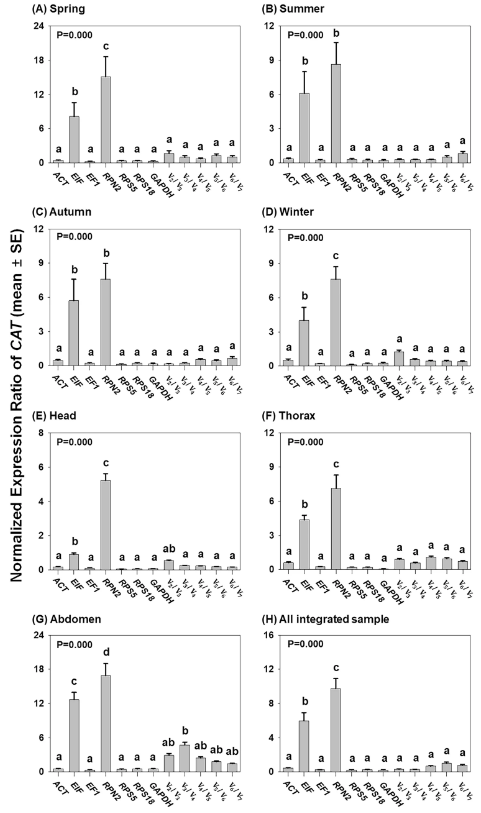
Валидация референтных генов
Поскольку парный анализ geNorm предполагает использование нескольких референсных генов для нормализации целевого гена (рис. 4), мы сравнили уровни экспрессии CAT (в качестве целевого гена), нормализованные каждым из семи референсных генов и несколькими референсными генами в течение четырех сезонов и трех типов тканей (рис. 5). При сезонном анализе количество выбранных референсных генов не изменило уровень экспрессии CAT (рис. 5A-D). Более того, уровни экспрессии КАТ, нормированные на один ген (т.е. ACT, EF1, RPS5, RPS18 или GAPDH), существенно не отличались от уровней экспрессии КАТ, нормированных на несколько референсных генов (P = 1,000 для весны, лета и осени; P = 0,868 для зимы) (рис. 5A-D). Эти результаты показывают, что один ген, а именно ACT, EF1, RPS5, RPS18 или GAPDH, может быть использован в качестве эталона при анализе сезонных тенденций экспрессии целевых генов у медоносных пчел. При сравнении экспрессии CAT в трех типах тканей, когда ACT, EF1, RPS5, RPS18 или GAPDH использовались как отдельные референсные гены, уровни экспрессии CAT существенно не отличались от уровней, полученных при анализе комбинации нескольких референсных генов в голове (P = 0,169), грудной клетке (P = 0,720) и брюшке (P = 0,379) (рис. 5E-G). Мы также сравнили общие уровни экспрессии CAT, нормализованные с одним референсным геном-кандидатом и комбинацией нескольких генов (рис. 5H). Анализ показал, что уровни экспрессии CAT, нормированные на любое количество комбинаций нескольких генов, существенно не отличались. Кроме того, при нормализации по ACT, EF1, RPS5, RPS18 и GAPDH уровни экспрессии CAT были статистически сходны с уровнями, полученными при комбинации нескольких генов (P = 0,981) (рис. 5H). Это говорит о возможности использования одного гена, ACT, EF1, RPS5, RPS18 или GAPDH, в качестве оптимального референсного гена для нормализации целевых генов в различные сезоны и типы тканей медоносной пчелы.
Обсуждение
Для того чтобы найти оптимальные референсные гены для qRT-PCR анализа у медоносной пчелы в течение четырех сезонов и трех типов тканей, мы оценили стабильность экспрессии семи кандидатных референсных генов: ACT, EIF, EF1, RPN2, RPS5, RPS18 и GAPDH, используя три программы анализа: NormFinder, BestKeeper и geNorm. Кроме того, анализ парных вариаций в geNorm был использован для определения оптимального количества референсных генов медоносной пчелы, необходимых для нормализации экспрессии целевых генов. Методы нормализации, включая отдельные эталонные гены или комбинации нескольких генов, также были проверены путем анализа экспрессии CAT в различных образцах.
В соответствии с наблюдениями предыдущих исследований (7,12,13), три алгоритма, NormFinder, BestKeeper и geNorm, дали разные результаты при ранжировании стабильности генов в настоящем исследовании; поэтому совместное использование всех алгоритмов обеспечило бы более достоверные результаты (13). Учитывая значения отсечки в каждом алгоритме, большинство из семи генов-кандидатов можно считать приемлемыми для использования в качестве эталонных генов при анализе экспрессии генов в различных условиях у медоносных пчел. Хотя в большинстве предыдущих исследований не было установлено значение отсечения для стабильности генов в анализе NormFinder (6,7,8,14,15,16,21,22), несколько последних исследований предложили значения < 0,15 в качестве подходящего отсечения (20,26,27). Исходя из этого критерия, все семь генов были подходящими референсными генами согласно NormFinder. Этот результат подтверждается распределением значений Cq, которое показало, что все семь генов-кандидатов стабильно экспрессировались со значениями CV < 1, что считается свидетельством низкой дисперсии (28). При анализе BestKeeper все семь генов также были определены как подходящие референсные гены для анализа экспрессии генов медоносной пчелы летом, осенью и среди трех типов тканей, на что указывают значения SD < 1 (22,23). Напротив, некоторые гены (ACT весной; RPN2, EF1 и ACT зимой; EF1 и ACT в интегрированном образце) не могут быть предложены в качестве оптимального эталонного гена из-за их значений SD > 1. При рассмотрении M ≤ 0,5 в качестве критерия для выбора подходящего референсного гена в анализе geNorm (21,24), все семь генов были приемлемыми референсами только в головке и интеграции всех образцов. Напротив, при использовании M ≤ 1,5 в качестве критерия, который был широко предложен в качестве приемлемого уровня для выбора эталонного гена (22,25), все семь генов могли рассматриваться в качестве эталонных генов по сезонам и типам тканей. В целом, наш комбинированный анализ показывает, что EIF, RPS5, RPS18 и GAPDH наиболее подходят в качестве оптимальных референсных генов для нормализации экспрессии целевых генов в образцах медоносных пчел, полученных из различных тканей в разные сезоны.
В дополнение к измерению стабильности экспрессии генов, geNorm может использоваться для дополнительного анализа значений парной вариации для возможного выбора нескольких референсных генов. В настоящем исследовании комбинация двух, трех, четырех, пяти или шести генов не повлияла на нормализацию целевых генов в трех типах тканей и интегрированном образце на основе Vn/Vn+1 < 0,15, что обычно использовалось в качестве отсекающего значения в анализе парной вариации geNorm в предыдущих исследованиях (21,22,25). Однако в разные сезоны значения V2/V3 для весны, осени и зимы были < 0,15, в то время как для летнего образца V5/V6 < 0,15, что показывает, что для нормализации экспрессии целевых генов весной, осенью и зимой рекомендуется использовать комбинацию из двух референсных генов, в то время как для летнего образца необходимо пять генов.
Независимо от оптимального количества референсных генов, предложенного для точной нормализации уровней экспрессии целевых генов с помощью парного анализа geNorm, V2/V3 предлагается в качестве минимального количества референсных генов; следовательно, по крайней мере, два гена необходимы в качестве внутреннего контроля, когда все значения Vn/Vn+1 < 0,15 (21,29). Однако для снижения финансовой и технической нагрузки в экспериментах может подойти выбор одного референсного гена, если уровни экспрессии целевых генов, полученные с одним референсным геном, существенно не отличаются от уровней, полученных с несколькими референсными генами (10,12). В настоящем исследовании, в разные сезоны и типы тканей, уровни экспрессии CAT с нормализацией по EIF или RPN2 были значительно выше, чем нормализованные по ACT, EF1, RPS5, RPS18 или GAPDH. Кроме того, значения Cq для EIF и RPN2 были относительно выше, чем для остальных пяти генов. Однако в разных условиях уровни экспрессии CAT при нормализации по ACT, EF1, RPS5, RPS18 или GAPDH были статистически сходны с уровнями экспрессии при использовании нескольких комбинаций референсных генов. Это говорит о том, что выбор одного референсного гена ACT, EF1, RPS5, RPS18 или GAPDH может быть возможной альтернативой комбинации нескольких референсных генов, несмотря на то, что парный анализ geNorm предполагает использование нескольких генов на основе значений Vn/Vn+1. Таким образом, при совместном анализе, каждый из ACT, EF1, RPS5, RPS18 и GAPDH может быть использован в качестве референсного гена для анализа методом qRT-PCR. Среди этих пяти генов, хотя другие анализы показали, что значения стабильности экспрессии всех пяти генов были ниже критериев, SD значения ACT и EF1 были больше 1,0, линии отсечения в BestKeeper. Поэтому, в заключение, все значения стабильности RPS5, RPS18 и GAPDH были ниже значений отсечки в каждом из использованных методов анализа. Таким образом, мы предполагаем, что RPS5, RPS18 и GAPDH являются наиболее подходящими референсными генами для точной нормализации экспрессии целевых генов в образцах медоносных пчел, полученных из различных тканей и сезонов.
Методы
Подготовка образцов и выделение тотальной РНК
Колонии медоносных пчел, использованные в данном исследовании (итальянский гибрид), содержались на экспериментальной пасеке Колледжа экологии и наук об окружающей среде Национального университета Кёнпук, Санджу, Кёнсанбук-до, Республика Корея (36° 22′ 24″ 41″ с.ш., 128° 08′ 24″ 24″ в.д.). Пчел-медсестер собирали из трех разных колоний весной (28 марта 2018 года), летом (27 июня 2017 года) и осенью (28 сентября 2017 года), исходя из возраста и поведения пчел; однако зимой (27 декабря 2017 года) пчел-медсестер получали из центральной области колонии, следуя предыдущему исследованию (5). Собранных медсестер немедленно замораживали жидким азотом и хранили при температуре — 70 °C до выделения РНК.
Для анализа тканей голову, грудную клетку и брюшко отделяли от пяти пчел и объединяли как одну репликацию в пробирку с реактивом TRI. Образцы РНК готовили из трех биологических реплик. Каждый образец ткани был полностью гомогенизирован с помощью блендера и общая РНК была выделена с помощью набора Direct-zol RNA Miniprep Plus (ZYMO RESEARCH, Ирвайн, Калифорния, США). Образцы обрабатывали ДНКазой I во время процедуры выделения РНК для устранения загрязнения геномной ДНК в соответствии с протоколом производителя (10). Чистоту и количество выделенной РНК измеряли в трех экземплярах с помощью спектрофотометра SpectraMax QuickDrop. Приготовленную РНК хранили при температуре — 70 °C до дальнейшего использования.
Конструирование праймеров и клонирование
Информация о последовательности семи генов была получена из базы данных NCBI (https://www.ncbi.nlm.nih.gov), а праймеры для семи референсных генов и целевого гена были разработаны в соответствии с предыдущим исследованием (7). Наборы праймеров для EF1, RPS18 и CAT были разработаны на основе двух разных экзонов для амплификации геномной ДНК, содержащей интроны, если образцы были загрязнены геномной ДНК; поэтому они давали более крупные продукты ПЦР (Таблица 1). Специфичность амплификации и отсутствие загрязнения геномной ДНК были подтверждены с помощью гель-электрофореза.
Для субклонирования тотальную РНК использовали в качестве шаблона для реакции ПЦР с обратной транскрипцией с помощью набора DiaStar OneStep RT-PCR (SOLGENT, Daejeon, Korea) по следующему протоколу: 50 °C в течение 30 мин; 95 °C в течение 15 мин; 35 циклов 95 °C в течение 20 с, 58 °C в течение 40 с и 72 °C в течение 30 с; и 5 мин при 72 °C. Для амплификации каждого гена использовались праймеры, специфичные для данного гена (Таблица 1). Затем их субклонировали в вектор pGEM-T easy (PROMEGA, Madison, MU, США). Плазмидосодержащие положительные вставки были представлены для реакции секвенирования с использованием универсальных праймеров M13 на анализаторе ABI PRISM 3730XL.
Количественная ПЦР в реальном времени
Для синтеза кДНК количество общей РНК стандартизировали до 1 мкг. Первая нить кДНК была синтезирована из тотальной РНК, выделенной из различных тканей в течение четырех сезонов с использованием обратной транскриптазы ReverTra Ace (TOYOBO, Осака, Япония), путем праймирования олиго (dT) в соответствии с протоколом производителя.
Для анализа qRT-PCR мы использовали систему обнаружения CFX Connect Real-Time PCR (BIO-RAD, Hercules, CA, USA) с методикой CYBR GREEN. Эффективность ПЦР каждого гена рассчитывали по заданному наклону после проведения стандартных кривых, построенных по четырем точкам двукратных серийных разведений кДНК по следующей формуле: E = 2-1/slope. Реакции qRT-PCR проводили в двух экземплярах (технические реплики) по следующему протоколу: 95 °C в течение 1 мин; затем 40 циклов 95 °C в течение 15 с, 58 °C в течение 15 с и 72 °C в течение 30 с. Значения цикла количественной оценки (Cq) семи референсных генов-кандидатов и целевого гена (CAT) были получены при одинаковом пороге флуоресценции (0,1).
Для валидации выбранных эталонных генов после определения их генной стабильности анализировали уровень экспрессии целевого гена (CAT). Значения Cq для референсных генов и CAT были получены для каждого образца и затем нормализованы методом относительной количественной оценки, адаптированным к оригинальной концепции 2-ΔΔCq (30). Референсные гены были выбраны на основе ранга стабильности генов, проанализированных с помощью geNorm (рис. 2), когда для нормализации использовалось несколько референсов, и для анализа использовалось среднее значение Cq.
Анализ данных
Распределение Cq генов по различным сезонам и тканям было проанализировано с помощью Origin Pro 9.0, были получены значения AM, SD и CV (CV = SD/AM).
Для оценки стабильности экспрессии семи кандидатных эталонных генов в данном исследовании использовались три свободно распространяемые программы, а именно NormFinder (версия 0.953) (20), BestKeeper (версия 1) (31) и geNorm (версия 3.1) (25). NormFinder рассчитывает значения стабильности каждого гена-кандидата на основе общей вариации для оценки систематической ошибки, вносимой при нормализации генов, при этом более низкие значения стабильности указывают на более стабильные гены (20). BestKeeper определяет подходящие эталонные гены, рассчитывая среднее геометрическое значение значений Cq генов, а затем SD: более низкие значения SD означают более стабильные гены. BestKeeper также рассчитывает корреляцию (R2) каждого гена-кандидата с другими генами. После этого высококоррелированные гены-кандидаты объединяются для оценки значений P. На основании результатов BestKeeper, гены-кандидаты с относительно высокими значениями R2, но низкими SD, CV и P считаются более стабильными генами. geNorm автоматически рассчитывает значение M для каждого предполагаемого эталонного гена на основе среднего геометрического значения всех исследованных генов: более стабильные гены обозначаются более низкими значениями M. Кроме того, geNorm сравнивает парную вариацию (V) гена с другими генами; парная вариация (Vn/Vn+1) рассчитывается для оценки оптимального количества референсных генов, необходимых для точной нормализации. Значение парной вариации ниже 0,15 указывает на то, что дополнительный референсный ген не улучшает нормализацию уровней экспрессии целевых генов (25).
SPSS for Windows (версия 23.0) был использован для статистического анализа экспрессии CAT (рис. 5). Характер экспрессии CAT между четырьмя сезонами и тремя типами тканей, нормированный на один ген или на несколько референсных генов, был статистически проанализирован с помощью одностороннего ANOVA с последующим пост-хок тестом множественного сравнения Тьюки (рис. 5A-H).
Дополнительная информация
Скачать Дополнительную информацию
Ссылки
1.Leonhardt, S. D., Gallai, N., Garibaldi, L. A., Kuhlmann, M. & Klein, A. M. Economic gain, stability of pollination and bee diversity decrease from southern to northern Europe. Basic Appl. Ecol. 14, 461–471. https://doi.org/10.1016/j.baae.2013.06.003 (2013).
Article Google Scholar
2.Lee, S. H. & Kim, Y. H. Comparative proteome analysis of honey bee workers between overwintering and brood-rearing seasons. J. Asia-Pac. Entomol. 20, 984–995. https://doi.org/10.1016/j.aspen.2017.07.011 (2017).
Article Google Scholar
3.Huang, Z. Y. & Robinson, G. E. Seasonal changes in juvenile hormone titers and rates of biosynthesis in honey bees. J. Comp. Physiol. B 165, 18–28. https://doi.org/10.1007/bf00264682 (1995).
PDF opens in a new tab Article CAS PubMed Google Scholar
4.Fluri, P., Lüscher, M., Wille, H. & Gerig, L. Changes in weight of the pharyngeal gland and haemolymph titres of juvenile hormone, protein and vitellogenin in worker honey bees. J. Insect Physiol. 28, 61–68.
https://doi.org/10.1016/0022-1910(82)90023-3 (1982). Article CAS Google Scholar
5.Kim, Y. H., Kim, J. H., Kim, K. & Lee, S. H. Expression of acetylcholinesterase 1 is associated with brood rearing status in the honey bee, Apis mellifera. Sci. Rep. 7, 39864. https://doi.org/10.1038/srep39864 (2017).
PDF opens in a new tab Article ADS CAS PubMed PubMed Central Google Scholar
6.Lourenco, A. P., Mackert, A., Cristino, A. D. & Simoes, Z. L. P. Validation of reference genes for gene expression studies in the honey bee, Apis mellifera, by quantitative real-time RT-PCR. Apidologie 39, 372–385. https://doi.org/10.1051/apido:2008015 (2008). Article CAS Google Scholar
7.Moon, K., Lee, S. H. & Kim, Y. H. Validation of quantitative real-time PCR reference genes for the determination of seasonal and labor-specific gene expression profiles in the head of Western honey bee, Apis mellifera. PLoS ONE 13, e0200369. https://doi.org/10.1371/journal.pone.0200369 (2018).
Article CAS PubMed PubMed Central Google Scholar
8.Zhai, Y. et al. Identification and validation of reference genes for quantitative real-time PCR in Drosophila suzukii (Diptera: Drosophilidae). PLoS ONE 9, e106800. https://doi.org/10.1371/journal.pone.0106800 (2014).
Article ADS CAS PubMed PubMed Central Google Scholar
9.Ling, D. & Salvaterra, P. M. Robust RT-qPCR data normalization: Validation and selection of internal reference genes during post-experimental data analysis. PLoS ONE 6, e17762. https://doi.org/10.1371/journal.pone.0017762 (2011).
Article ADS CAS PubMed PubMed Central Google Scholar
10.Moon, K., Lee, S. H. & Kim, Y. H. Evaluation of reference genes for quantitative real-time PCR to investigate seasonal and labor-specific expression profiles of the honey bee abdomen. J. Asia-Pac. Entomol. 21, 1350–1358. https://doi.org/10.1016/j.aspen.2018.10.014 (2018).
Article Google Scholar
11.Kim, Y. et al. Validation of reference genes for quantitative real-time polymerase chain reaction in Drosophila melanogaster exposed to two chemicals. Entomol. Res. 49, 277–283. https://doi.org/10.1111/1748-5967.12365 (2019).
PDF opens in a new tab Article CAS Google Scholar
12.Kim, Y., Kim, Y. & Kim, Y. H. Evaluation of reference genes for gene expression studies using quantitative real-time PCR in Drosophila melanogaster after chemical exposures. J. Asia-Pac. Entomol. 23, 385–394. https://doi.org/10.1016/j.aspen.2020.01.008 (2020). Article Google Scholar
13.Wang, Z. et al. Evaluation and validation of reference genes for quantitative real-time PCR in Helopeltis theivora Waterhouse (Hemiptera: Miridae). Sci. Rep. 9, 13291. https://doi.org/10.1038/s41598-019-49479-1 (2019).
PDF opens in a new tab Article ADS CAS PubMed PubMed Central Google Scholar
14.Ponton, F., Chapuis, M. P., Pernice, M., Sword, G. A. & Simpson, S. J. Evaluation of potential reference genes for reverse transcription-qPCR studies of physiological responses in Drosophila melanogaster. J. Insect Physiol. 57, 840–850. https://doi.org/10.1016/j.jinsphys.2011.03.014 (2011). Article CAS PubMed Google Scholar
15.Scharlaken, B. et al. Reference gene selection for insect expression studies using quantitative real-time PCR: The head of the honeybee, Apis mellifera, after a bacterial challenge. J. Insect. Sci. 8, 1–10. https://doi.org/10.1673/031.008.3301 (2008).
PDF opens in a new tab Article Google Scholar
16.Reim, T., Thamm, M., Rolke, D., Blenau, W. & Scheiner, R. Suitability of three common reference genes for quantitative real-time PCR in honey bees. Apidologie 44, 342–350. https://doi.org/10.1007/s13592-012-0184-3 (2013).
PDF opens in a new tab Article CAS Google Scholar
17.Antunez, K. et al. Immune suppression in the honey bee (Apis mellifera) following infection by Nosema ceranae (Microsporidia). Environ. Microbiol. 11, 2284–2290. https://doi.org/10.1111/j.1462-2920.2009.01953.x (2009). Article CAS PubMed Google Scholar
18.McQuillan, H. J., Barron, A. B. & Mercer, A. R. Age- and behaviour-related changes in the expression of biogenic amine receptor genes in the antennae of honey bees (Apis mellifera). J. Comp. Physiol. A Neuroethol. Sens. Neural. Behav. Physiol. 198, 753–761. https://doi.org/10.1007/s00359-012-0745-y (2012).
PDF opens in a new tab Article CAS PubMed Google Scholar
19.Richard, F. J., Schal, C., Tarpy, D. R. & Grozinger, C. M. Effects of instrumental insemination and insemination quantity on Dufour’s gland chemical profiles and vitellogenin expression in honey bee queens (Apis mellifera). J. Chem. Ecol. 37, 1027–1036. https://doi.org/10.1007/s10886-011-9999-z (2011).
PDF opens in a new tab Article CAS PubMed Google Scholar
20.Andersen, C. L., Jensen, J. L. & Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64, 5245–5250. https://doi.org/10.1158/0008-5472.CAN-04-0496 (2004).
Article CAS PubMed Google Scholar
21.Liu, C. H. et al. Reference gene selection for quantitative real-time RT-PCR normalization in the half-smooth tongue sole (Cynoglossus semilaevis) at different developmental stages, in various tissue types and on exposure to chemicals. PLoS ONE 9, e91715. https://doi.org/10.1371/journal.pone.0091715 (2014).
Article ADS CAS PubMed PubMed Central Google Scholar
22.Wan, H. J. et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber. Anal. Biochem. 399, 257–261. https://doi.org/10.1016/j.ab.2009.12.008 (2010).
Article CAS PubMed Google Scholar
23.Chechi, K., Gelinas, Y., Mathieu, P., Deshaies, Y. & Richard, D. Validation of reference genes for the relative quantification of gene expression in human epicardial adipose tissue. PLoS ONE 7, e32265. https://doi.org/10.1371/journal.pone.0032265 (2012).
Article ADS CAS PubMed PubMed Central Google Scholar
24.Hellemans, J., Mortier, G., De Paepe, A., Speleman, F. & Vandesompele, J. qBase relative quantification framework and software for management and automated analysis of real-time quantitative PCR data. Genome Biol. 8, R19. https://doi.org/10.1186/gb-2007-8-2-r19 (2007). Article CAS PubMed PubMed Central Google Scholar
25.Vandesompele, J. et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. https://doi.org/10.1186/gb-2002-3-7-research0034 (2002).
Article PubMed PubMed Central Google Scholar
26.Julian, G. S., Oliveira, R. W., Tufik, S. & Chagas, J. R. Analysis of the stability of housekeeping gene expression in the left cardiac ventricle of rats submitted to chronic intermittent hypoxia. J. Bras. Pneumol. 42, 211–214. https://doi.org/10.1590/S1806-37562015000000133 (2016). Article PubMed PubMed Central Google Scholar
27.McMillan, M. & Pereg, L. Evaluation of reference genes for gene expression analysis using quantitative RT-PCR in Azospirillum brasilense. PLoS ONE 9, e98162. https://doi.org/10.1371/journal.pone.0098162 (2014).
Article ADS CAS PubMed PubMed Central Google Scholar
28.Ospina, R. & Marmolejo-Ramos, F. Performance of some estimators of relative variability. Front. Appl. Math. 5, 00043. https://doi.org/10.3389/fams.2019.00043 (2019). Article Google Scholar
29.Reid, K. E., Olsson, N., Schlosser, J., Peng, F. & Lund, S. T. An optimized grapevine RNA isolation procedure and statistical determination of reference genes for real-time RT-PCR during berry development. BMC Plant Biol. 6, 27. https://doi.org/10.1186/1471-2229-6-27 (2006). PDF opens in a new tab Article CAS PubMed PubMed Central Google Scholar
30.Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 29, e45. https://doi.org/10.1093/nar/29.9.e45 (2001).
PDF opens in a new tab Article CAS PubMed PubMed Central Google Scholar
31.Pfaffl, M. W., Tichopad, A., Prgomet, C. & Neuvians, T. P. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations. Biotechnol. Lett. 26, 509–515. https://doi.org/10.1023/b:bile.0000019559.84305.47 (2004).
Article CAS PubMed Google Scholar
Добавить комментарий